 Claudine Junien use depuis deux ans de tout son poids de professeure émérite de génétique et membre de l’Académie nationale de médecine pour diffuser l’idée suivante : la « différence génétique » entre hommes et femmes est égale à 1.5%, une différence selon elle quinze fois plus grande que celle existant en moyenne entre deux hommes, et comparable à celle existant entre le chimpanzé et l’humain. Comme on va le voir, le calcul de ce pourcentage est plus que spécieux dans son principe et complètement erroné dans sa mise en œuvre, et ces deux comparaisons n’ont aucun sens. La mobilisation de cette rhétorique fallacieuse ne peut s’expliquer autrement que par une volonté de défendre à tout prix ce qu’elle appelle « la cause du sexe biologique », ce qui n’est pas sans rappeler certains précédents.
Claudine Junien use depuis deux ans de tout son poids de professeure émérite de génétique et membre de l’Académie nationale de médecine pour diffuser l’idée suivante : la « différence génétique » entre hommes et femmes est égale à 1.5%, une différence selon elle quinze fois plus grande que celle existant en moyenne entre deux hommes, et comparable à celle existant entre le chimpanzé et l’humain. Comme on va le voir, le calcul de ce pourcentage est plus que spécieux dans son principe et complètement erroné dans sa mise en œuvre, et ces deux comparaisons n’ont aucun sens. La mobilisation de cette rhétorique fallacieuse ne peut s’expliquer autrement que par une volonté de défendre à tout prix ce qu’elle appelle « la cause du sexe biologique », ce qui n’est pas sans rappeler certains précédents.
C’est semble-t-il dans la bouche du généticien états-unien Huntington Willard, selon Sarah Richardson (2010), qu’est née en 2005 l’estimation d’une différence génétique entre femmes et hommes comprise entre 1 et 2%, ainsi que l’idée que cette distance génétique était comparable à celle nous séparant du chimpanzé. Il s’exprimait alors dans le cadre de la promotion d’un travail de recherche qu’il venait de publier avec Laura Carrel dans Nature. Cette estimation et cette comparaison faisant des hommes et des femmes deux groupes aussi différents que deux espèces ayant chacune son génome – absentes de l’article scientifique – ont alors été diffusées dans l’espace public anglo-saxon.
En France, c’est dix ans plus tard que cette rhétorique a fini par sortir de la confidentialité. Suggérée par la « journaliste scientifique » Peggy Sastre avant d’être développée et légitimée par la généticienne Claudine Junien (avec un petit coup de pouce de Nicolas Gauvrit, mathématicien et chercheur en mathématiques appliquées à la psychologie cognitive), elle est activement mobilisée par les mêmes pour défendre l’idée que les différences biologiques liées au sexe sont à l’origine de différences essentielles entre femmes et hommes, y compris (voire surtout) des différences psychologiques.
Je présente dans ce qui suit une histoire du développement de cette rhétorique, et montre pourquoi elle est multiplement et profondément fallacieuse. Les lecteur·ices déjà bien informé·es ou simplement pressé·es pourront sauter directement à la deuxième partie, voire à la troisième et dernière partie en utilisant le plan ci-dessous. Pour les autres, j’expose dans une première partie les éléments de génétique nécessaires à la bonne compréhension de la suite.
Au-delà du contexte de la critique des propos de Claudine Junien, j’espère que cette première partie sera plus généralement utile aux personnes qui en sont restées aux idées prévalant dans les années 1990, et qui les transmettent pour certaines encore aujourd’hui à leurs élèves. Mon fils en 3ème cette année vient ainsi d’apprendre en cours de SVT que « l’information génétique est l’information héréditaire portée par l’ensemble des gènes sur les chromosomes », qu’un gène est « une portion d’ADN qui détermine un caractère », que le « patrimoine » génétique de l’être humain contient « 30 000 gènes », et que nous avons « 98.8% de notre information génétique en commun avec le chimpanzé » [1]. Comme on va le voir, tout cela est faux.
_____________________
Plan
PARTIE 1 – PREREQUIS DE GENETIQUE
Vocabulaire et notions de base
Caryotypes et spécificités du X et du Y chez l’humain et le chimpanzé
Zoom sur la région spécifique au chromosome Y humain
Nombre de gènes et taille du génome humain
Eléments de génomique comparative
Variabilité génétique humaine
Signification fonctionnelle d’un pourcentage de différence entre génomes humains
Différence génomique entre humains et chimpanzés
PARTIE 2 – BREVE HISTOIRE DE L’ARGUMENT DE CLAUDINE JUNIEN
Début des années 2000 : Mattei, Ginger et compagnie
2005 : Carrel et Willard publient des données à prendre avec des pincettes
Publicité/communication des résultats de Carrel et Willard
Relais fantasque de l’étude de Carrel et Willard dans les médias anglo-saxons
Relais plus factuel dans les médias français, faux et trompeur par Peggy Sastre
2006 : mise en question de la portée des résultats de Carrel et Willard
2008 : confirmation de la portée limitée des résultats de Carrel et Willard
Peggy Sastre enfonce néanmoins le clou, puis Claudine Junien arrive en renfort
Le fameux argument arrive dans la bouche de Claudine Junien
Déploiement de l’argument choc
PARTIE 3 – POURQUOI C’EST MULTIPLEMENT FAUX ET INEPTE
Calcul de la « différence génétique » entre femmes et hommes
Prise en compte des niveaux d’expression
Comparaison avec la différence entre deux humains de même sexe
Comparaison avec la différence entre humain et chimpanzé
PARTIE 1 – PREREQUIS DE GENETIQUE
Vocabulaire et notions de base
Chez l’humain comme chez le chimpanzé, l’information génétique contrôlant le fonctionnement des cellules est portée par des molécules d’acide désoxyribonucléique (ADN). L’ADN est contenu en quasi-totalité par les chromosomes présents dans le noyau des cellules (ADN nucléaire), mais il en existe également une petite partie dans les mitochondries, des organites présents dans le cytoplasme des cellules (ADN mitochondrial).
Au départ, le bagage génétique d’un humain ou d’un chimpanzé est constitué d’un mélange de celui présent dans l’ovule de sa génitrice et de celui présent dans le spermatozoïde de son géniteur, chacun correspondant en principe à environ la moitié du bagage génétique présent dans leurs cellules somatiques respectives. Cependant, il arrive que l’ADN d’un gamète comporte une variante génétique absente des cellules somatiques, dont l’individu « hérite » alors bien qu’elle soit absente du bagage génétique de ses géniteurs (on parle dans ce cas de « mutation de novo »).
En dehors des mutations aléatoires ou des erreurs de copie pouvant affecter certaines lignées cellulaires au cours de la vie (ou beaucoup plus rarement toutes les cellules de l’individu, lorsque cela se produit juste après la formation de l’œuf qui est à son origine), toutes les cellules somatiques d’un individu sont en principe dotées du même bagage génétique. Dans le cas contraire, appelé chimérisme, très rare chez l’humain au moins, l’individu est formé de plusieurs populations de cellules génétiquement distinctes, typiquement suite à la fusion très précoce de deux œufs.
Les molécules d’ADN sont faites de très longs brins constitués d’une succession de ce qu’on appelle des nucléotides, chaque nucléotide contenant une base azotée. Ces bases azotées sont de quatre types différents, dont les initiales des noms sont A, C, G et T. De ce fait, l’information génétique portée par l’ADN peut être vue comme un code écrit à l’aide de ces quatre lettres. La longueur d’un morceau d’ADN est exprimée en nombre de bases (ce qui équivaut donc à un nombre de nucléotides ou à un nombre de « lettres » dans le « code génétique »), deux unités de mesure courantes étant le kilobase (kb) et le mégabase (Mb), égaux à 1000 bases et 1 million de bases respectivement.
Un gène est une séquence d’ADN susceptible d’être transcrite en ARN. L’ARN est une séquence de nucléotides qui est la copie du code de l’ADN source, avec simplement des ‘U’ à la place des ‘T’. Si l’ARN produit par la transcription d’un gène est de l’ARN messager (ARNm), il sera exporté vers le cytoplasme de la cellule pour être traduit en protéine, et le gène est dit « codant pour une protéine », ou plus simplement codant. Sinon, le gène est dit non codant, et l’ARN produit peut jouer un rôle dans la régulation de la transcription de l’ADN, la maturation des ARN (voir plus bas) ou encore la maintenance du génome, ou bien n’avoir aucune fonction. On pense en effet qu’il existe une sorte de « bruit transcriptionnel » à l’intérieur des cellules, à savoir une transcription aléatoire et en très petite quantité de portions de l’ADN ne jouant aucun rôle biologique.
On trouve aussi dans la partie non codante de l’ADN ce qu’on appelle des pseudogènes, qui sont des reliquats de gènes devenus non fonctionnels à cause de mutations délétères, ou qui proviennent de la réinsertion accidentelle dans le génome de séquences d’ADN issues d’une transcription inverse de brins d’ARNm. Les pseudogènes sont réputés inactifs, mais ils ne sont pas nécessairement dépourvus de fonction pour autant. On a ainsi observé que dans certains cas, un pseudogène pouvait agir comme un leurre vis-à-vis de mécanismes biologiques empêchant l’expression d’un gène auquel il ressemble : ils sont pris pour cible en même temps que ce dernier, qui de ce fait est relativement « épargné ».
Après la transcription en ARN d’une séquence d’ADN (notamment si elle est codante, mais cela arrive pour certaines non codantes), des parties appelées introns sont excisées pour ne laisser que ce qu’on appelle les exons, et ceux-ci sont ensuite « collés » ensemble pour former l’ARN mature. Ce processus d’excision/collage après transcription est appelé épissage. L’épissage ne respecte pas toujours l’ordre initial des exons et ne les inclut pas toujours tous dans l’ARN mature : le cas échéant, l’épissage est dit alternatif. La succession des codons (séquences de trois nucléotide) d’un ARNm mature est ainsi déterminée non seulement par la succession des bases du gène mais aussi par l’épissage, et cette succession des codons détermine ensuite celle des acides aminés constituant la protéine qui sera issue de la traduction de l’ARNm. Du fait des possibilités d’épissage alternatif, un même gène codant peut donc coder pour plusieurs protéines différentes, et la composition finale des protéines formées par la traduction de l’ARNm dépend en outre d’autres facteurs de l’environnement de la cellule. Une protéine donnée intervient ensuite, souvent en intéraction avec d’autres, dans le développement ou le maintien d’un ou plusieurs « caractères » de l’individu, l’ensemble de ceux-ci étant appelé son phénotype.
Un gène est dit exprimé lorsque l’information qu’il contient est utilisée pour la synthèse de son produit final. Le niveau d’expression d’un gène non codant est mesuré par la quantité de l’ARN issu de sa transcription, et celui d’un gène codant est idéalement mesuré par la quantité produite de protéine(s) correspondante(s), mais est souvent estimé indirectement par la mesure de la quantité d’ARNm issu de sa transcription. Le niveau d’expression d’un gène peut être mesuré à l’aide de différentes techniques, donnant des résultats pouvant être différents, et cela nécessite des manipulations complexes effectuées sur des cellules extraites du corps.
Au sein d’un individu, tous les gènes ne sont pas exprimés dans toutes les cellules, et l’épissage ainsi que le niveau d’expression d’un gène sont également variables. L’expression d’un gène dépend de l’environnement physico-chimique interne à la cellule et des signaux qu’elle reçoit, et est soumise à des régulations complexes opérées par d’autres parties de l’ADN, ainsi que par des molécules présentes autour de l’ADN. L’expression d’un gène dans une cellule donnée dépend ainsi non seulement du reste du bagage génétique de l’individu et de la localisation de la cellule dans son corps, mais aussi de son histoire développementale et de ses interactions avec son environnement.
Caryotypes et spécificités du X et du Y chez l’humain et le chimpanzé
Les êtres humains sont la plupart du temps dotés de 46 chromosomes formant 23 paires : les paires d’autosomes (chromosomes semblables) numérotés de 1 à 22, et une paire de chromosomes pouvant être différents (hétérochromosomes), appelés X et Y chez les mammifères. Notre génome est dit diploïde, fait de la réunion de deux génomes dit haploïdes, l’un hérité de notre génitrice et l’autre de notre géniteur, chaque génome haploïde contenant en principe une copie des 22 autosomes et un hétérochromosome. L’ADN mitochondrial est quant à lui hérité uniquement de la génitrice.
Les personnes ordinairement appelées « femmes » ont le plus souvent un caryotype 46,XX, soit 46 chromosomes dont deux X, et celles appelées « hommes » un caryotype 46,XY. Les chimpanzés, dont le génome est également diploïde, sont quant à eux en principe dotés de 48 chromosomes formant 24 paires : les paires d’autosomes numérotés 1, 2a, 2b, puis 3 à 22, et une paire d’hétérochromosomes. Une femelle chimpanzé a en principe un caryotype 48,XX et un mâle un caryotype 48,XY.
Dans les deux espèces, les gènes du chromosome Y sont répartis en deux groupes : ceux des régions pseudo-autosomiques (PAR), et ceux de la région « spécifique aux mâles » (MSY), autrefois appelée région non recombinante (NRY). A l’instar des autosomes, les régions PAR1 et PAR2 situées aux extrémités du Y peuvent se recombiner avec les régions correspondantes du chromosome homologue – en l’occurrence le X – lors de la production des spermatozoïdes. Les gènes des régions PAR, dont 19 gènes codants selon HGNC (11/2017), sont donc les mêmes sur le Y et sur le X. En revanche, la région MSY a perdu sa capacité de recombinaison avec le X et son évolution a de ce fait divergé de celle du X. Accumulant les mutations défavorables, elle a perdu une grande partie des gènes ancestraux (97% chez l’humain selon Bellott et al. 2014). Le X s’est quant à lui bien maintenu, car les deux X se recombinent lors de la production des ovocytes. Par ailleurs, de nouveaux gènes sont apparus sur le X et le Y au cours de l’évolution. Ils semblent particulièrement impliqués dans le fonctionnement des testicules (Bellott et al. 2014), certains gènes portés par le X ne s’exprimant même que dans ceux-ci.
Comme ceux des autosomes, les gènes portés par le chromosome X ont des fonctions très diverses et s’expriment plus ou moins selon les tissus. Par ailleurs, le chromosome X contient un gène non codant très important appelé Xist. Il s’exprime systématiquement sur l’un des deux chromosomes X présents dans les cellules XX (de même que sur tout chromosome X surnuméraire dans le cas où l’individu possède deux X et un Y, ou trois X ou plus, ou encore deux X et deux Y). Son expression a pour effet de faire se recroqueviller le chromosome X qui le porte, empêchant une grande partie de ses gènes de s’exprimer. Le X concerné est alors dit inactivé bien que certains de ses gènes s’expriment, notamment ceux des régions PAR.
Zoom sur la région spécifique au chromosome Y humain
Il reste dans la région MSY des gènes homologues à des gènes du X, c’est-à-dire issus d’un gène ancestral commun, mais qui sont devenus différents. C’est notamment le cas du gène Sry, dont l’homologue sur le X est appelé Sox3. Sa présence est un facteur clé de la sexuation biologique : toutes choses égales par ailleurs, si l’environnement et le reste du bagage génétique sont habituels, l’individu développe des testicules s’il est porteur du gène Sry, et des ovaires sinon.
Selon l’étude approfondie de la région MSY humaine publiée par Jangravi et al. (2013), celle-ci comprend environ 95% de la longueur du Y, mais seulement « environ 40 » gènes codant pour des protéines (44 selon HGNC 11/2017). Parmi eux, 29 semblent ne s’exprimer que dans les testicules, les spermatozoïdes ou leurs précurseurs [2]. Sur les 63 gènes codants actuellement recensés sur le Y humain (Ensemble v90 ; HGNC 11/2017), seuls 44 sont donc spécifiques au Y, et parmi eux seulement 15 semblent s’exprimer ailleurs que dans les testicules et les gamètes masculins.
Jangravi et al. (2013) passent en revue les liens identifiés ou suggérés par des études publiées antérieurement entre les gènes de la région MSY et des traits ou pathologies. Ces liens concernent le développement des testicules et leur fonctionnement (spermatogénèse, tumeur des cellules germinales…) ainsi que la prostate. Seules quatre mentions relatives à des gènes de la région MSY concernent d’autres parties du corps.
La première concerne le gène KDM5D. Il code pour un précurseur de l’antigène H-Y, qui est une source potentielle de réaction du système immunitaire. Cet antigène semble être impliqué dans le risque plus élevé de rejet d’une greffe de moelle épinière par une femme quand le donneur est un homme. Il pourrait également induire un ciblage parfois délétère de certaines cellules des fœtus masculins par les lymphocytes T de leur gestatrice (Linscheid et Petroff 2013).
La seconde concerne le gène PCDH11Y, qui comme PCDH11X, son homologue sur le X (à 98.1% selon les auteur·es), a un haut niveau d’expression dans la moelle épinière et le cerveau des fœtus selon les auteur·es (référence citée : Yoshida et Sugano 1999). Les auteur·es évoquent uniquement l’observation du cas d’un garçon dont les gènes PCDH11X et PCDH11Y portaient tous deux une délétion et qui présentait un retard de langage. Il avait été postulé que cela altérait chez lui l’épissage et par conséquent l’expression de ces deux gènes. Les auteur·es soulignent que ces deux gènes sont absents chez le chimpanzé, « ce qui étaye l’hypothèse selon laquelle ils ont pu jouer un rôle significatif dans la transition vers le développement du langage et du cerveau des humains modernes » (p. 13).
La troisième concerne les gènes TBL1Y et NLGN4Y, dont une analyse comparée des variantes génétiques trouvées chez 146 autistes et 102 contrôles « d’origine euro-américaine » a « suggéré » un rôle dans les troubles autistiques (référence citée : Serajee et Hug 2009), ce qui pourrait contribuer à la prévalence plus grande de l’autisme observée chez les garçons que chez les filles. Les auteur·es signalent que la contribution du chromosome Y au développement de l’autisme est également suggérée par le fait que les personnes de caryotype 47,XYY ont « souvent un phénotype autistique », ainsi qu’environ 30% de celles de caryotype 48,XXYY selon une étude menée sur 95 d’entre elles (références citées : Fryns et al. 1995 et Tartaglia et al. 2009).
La quatrième et dernière mention concerne plusieurs gènes de la région MSY (dont USP9Y, DDX3Y, RPS4Y1 et EIF1AY) qu’on a trouvé nettement « surexprimés » dans le muscle cardiaque chez des hommes ayant eu un infarctus et atteints d’une maladie du muscle cardiaque particulière, causant une dilatation des cavités du cœur. Selon les auteur·es, les gènes spécifiques au Y pourraient ainsi être à l’origine de différences entre femmes et hommes dans les troubles cardiaques. Cela pourrait aussi être le cas du gène Xist, qui si l’on en croit l’étude citée (Heidecker et al. 2010) est hautement surexprimé chez les femmes de leur échantillon atteintes de la même pathologie.
Nombre de gènes et taille du génome humain
Bien que l’« achèvement » de son séquençage ait été annoncé en 2004 (International Human Genome Sequencing Consortium 2004), la description du génome humain est un travail toujours en cours. Outre que sa variabilité au sein de l’espèce n’est que partiellement documentée, certaines de ses portions complexes restent mal décrites, et l’identification de tous les gènes reste en cours. En effet, on ne sait pas directement « décrypter » l’ADN, c’est-à-dire le « lire » en déterminant avec certitude le type de chaque séquence identifiée. La construction d’un génome de référence et la catégorisation des séquences qui le constituent est un processus très complexe, en partie fondé sur des calculs de probabilités, et la distinction entre gènes codants, gènes non codants et pseudogènes n’est pas toujours aisée. Kim et al. (2014) ont ainsi trouvé que 140 séquences référencées comme étant des pseudogènes, 9 gènes référencés comme non codants et 34 autres morceaux d’ADN censés ne pas être codants semblaient pourtant coder pour des protéines.
De ce fait, même le nombre de gènes codants fait encore l’objet d’estimations variables. Leur nombre estimé tend à diminuer au fil du temps : de « environ 30 000 » en 2001, il est ainsi passé à « 20 000 à 25000 » en 2004, puis environ 21 000 en 2007, l’estimation actuelle la plus consensuelle étant d’environ 20 300 (Ensembl 2017). Ce nombre pourrait toutefois encore baisser notablement, car pour environ 15 % des gènes jugés codants, il manque encore la preuve expérimentale de la production effective de protéines issues de leur transcription (selon www.humanproteomemap.org accédé en novembre 2017, données issues de Kim et al. 2014). Par ailleurs, on ne sait pas quel pourcentage de l’ADN non codant (qui représente plus de 98 % de notre génome) exerce une influence biologique. Son estimation fait débat, allant de 10 % jusqu’à plus de 80 % selon certain·es chercheur·es.
Dans la base de référence Ensembl, le décompte du nombre total de gènes est basé sur un génome haploïde sauf pour les hétérochromosomes, et non redondant sauf pour les régions PAR, auquel est ajouté l’ADN mitochondrial. En d’autres termes, les gènes du X et du Y sont tous deux comptés, et ceux des régions PAR, communs aux deux hétérochromosomes, sont comptés à la fois sur le X et sur le Y. En revanche, un gène porté par une paire d’autosomes n’est compté qu’une fois, de même qu’un gène dont plusieurs copies peuvent éventuellement être présentes les unes à la suite des autres sur un chromosome donné. Il faut savoir que de telles duplications de gènes sont fréquentes dans le génome humain, et peuvent inclure un grand nombre de copies. Par exemple, 6 gènes de la famille TSPY sont recensés dans la région MSY humaine (HGNC 11/2017), mais 27 à 40 exemplaires des gènes de cette famille sont habituellement présents chez les porteurs du chromosome Y (Jangravi et al 2013).
Avec ce mode de calcul, Ensembl v90 (2017) décompte 20 309 gènes codants : 19 392 sur les autosomes, 841 sur le X et 63 sur le Y (dont 19 dans les régions PAR selon HGNC 11/2017), et 13 dans l’ADN mitochondrial. Malgré ce petit nombre, plus de 200 000 transcrits de gènes codants différents sont répertoriés, la plupart des gènes humains étant sujets à épissage alternatif. Les gènes non codants sont quant à eux estimés à 22 281 : 21 510 sur les autosomes, 639 sur le X et 108 sur le Y (dont 8 dans les régions PAR selon HGNC 11/2017) et 24 dans l’ADN mitochondrial, et les pseudogènes à 14 632 : 13 369 sur les autosomes, 872 sur le X et 391 sur le Y (dont 8 dans les régions PAR selon HGNC 11/2017), et aucun dans l’ADN mitochondrial. Le tableau ci-après synthétise ces décomptes et reconstitue des totaux en évitant le comptage en double des gènes de PAR1 et PAR2.
En ce qui concerne la longueur totale du génome selon Ensembl v90 (2017), elle est d’environ 3106 Mb : 2897 Mb sur les autosomes, 156 Mb sur le X, 57 Mb sur le Y et moins de 17 kb dans l’ADN mitochondrial.
Eléments de génomique comparative
Les différences entre deux génomes peuvent être issues de l’accumulation de mutations ponctuelles ou de réarrangements génomiques de plus grande ampleur intervenus au cours de leur histoire. Dans une perspective évolutive, ce qu’on appelle habituellement une mutation est soit la substitution d’un nucléotide à un autre (une « lettre » est changée), soit la délétion ou l’insertion d’un nucléotide au sein d’une séquence d’ADN (suppression ou ajout d’une « lettre »). Lorsqu’on compare les génomes d’individus de la même espèce, si une base présente au sein d’une séquence donnée du génome de référence fait parfois l’objet d’une substitution par une autre, cette variation est appelée un SNP (pour single nucleotide polymorphism).
Les réarrangements génomiques incluent la délétion ou l’insertion d’un segment d’ADN, l’inversion de l’ordre d’une suite de segments, le déplacement d’un segment sur une autre partie du même chromosome (transposition), et la recopie de segments sur le même chromosome (duplication). Lorsqu’on compare les génomes d’individus de la même espèce, si un segment d’au moins 50b de long est parfois dupliqué (une ou plusieurs fois) ou au contraire absent chez certains, cette variation est appelée un CNV (pour copy-number variant). La figure ci-après représente les grands types de réarrangements génomiques intrachromosomiques et leur traduction en termes de variabilité structurelle lorsqu’on compare des génomes d’individus de la même espèce.
Les réarrangements génomiques peuvent également impliquer plusieurs chromosomes : échange de segments entre deux chromosomes (translocation), fusion de deux chromosomes, ou au contraire fission d’un chromosome. Ainsi, le chromosome 2 humain est manifestement issu de la fusion de deux chromosomes qui étaient présents chez notre dernier ancêtre commun avec le chimpanzé, qui sont restés séparés chez ce dernier – d’où la numérotation 2a et 2b adoptée pour ceux-ci.
Compte tenu de toutes ces possibilités, ainsi que de la combinaison possible de plusieurs modifications portant sur un site donné du génome (ex : duplication d’une séquence dont certaines bases sont en outre substituées, translocation d’une séquence en outre inversée, etc), la comparaison de deux génomes est un exercice complexe. Outre ses nombreuses difficultés techniques, cet exercice nécessite l’emploi de logiques différentes selon les aspects du génome que l’on souhaite comparer, et la quantification de la différence entre deux génomes dépend de la logique adoptée.
Une étape préalable est l’alignement des deux génomes, qui consiste à retrouver les séquences d’ADN qui se correspondent « assez bien » à condition d’autoriser des « petits » trous, ces séquences étant présumées avoir un ancêtre commun ayant subi des mutations. Après alignement de ces séquences deux à deux, on peut quantifier les substitutions de nucléotides et les « petites » insertions/délétions (appelées indels) existant en leur sein. La figure ci-après illustre le principe général de la démarche.
La comparaison en termes de « petites » différences entre deux séquences alignées ne quantifie que l’un des aspects de la différence entre les deux génomes, puisqu’elle exclut par construction les séquences non alignées et ne tient pas compte des différences de plus grande ampleur induites par des réarrangements génomiques (duplications, translocations,…).
Lorsqu’on quantifie les différences entre génomes de deux espèces, c’est en général dans une perspective de biologie évolutive, pour estimer le temps écoulé depuis leur divergence et tenter de reconstituer leurs histoires évolutives respectives. Dans ce cadre, on s’intéresse séparément à tel ou tel type de région du génome (ex : portions codantes vs non codantes ; autosomes vs régions non recombinantes des hétérochromosomes ; chromosome Y vs ADN mitochondrial) et tel ou tel type de différences entre eux (ex : substitutions de nucléotides vs indels de grande taille). En fonction de ces choix, le pourcentage de différence varie, ainsi que le type d’information apportée par ce calcul. Il en est de même lorsqu’on quantifie la différence entre génomes de plusieurs populations humaines dans le cadre des recherches en génétique des populations.
Une partie du génome qui est variable au sein de l’espèce, c’est-à-dire présentant des variantes différentes selon les individus, est appelée un polymorphisme. Dans le contexte de l’étude de la variabilité génétique humaine, une variante génétique trouvée chez moins de 1% des personnes, ce qui est une anomalie statistique selon le seuil conventionnel habituel, est communément appelée une mutation. Cette distinction conventionnelle est peut-être appelée à disparaître car une variante rare au sein d’une population donnée peut l’être moins dans une autre, et aussi parce que cet emploi du mot mutation est source de confusions : il conviendrait de le réserver aux modifications apparaissant au cours de l’histoire d’une lignée d’individus ou de cellules (Karki et al. 2015).
Tous les grands types de variations génétiques existent au sein de l’espèce humaine, y compris des variations très macroscopiques en termes de caryotype. Selon Genetics Home Reference (2017), les plus fréquentes parmi ces dernières sont le caryotype 47,XXY (entre une 0.1 et 0.2 % des personnes), la trisomie 21 (environ 0.125 %), les caryotypes 47,XXX et 47,XYY (environ 0.05 % chacun) et le caryotype 45,X0 (environ 0.02 %). Par ailleurs, certaines personnes XX ont un morphotype plutôt masculin (typiquement en cas de translocation du gène Sry sur le X ou sur un autosome), certaines personnes XY ont un morphotype plutôt féminin (typiquement en cas de possession d’une mutation génétique les rendant complètement insensibles aux androgènes), et d’autres encore ont un morphotype ambigu. La possession d’organes génitaux ambigus à la naissance, souvent estimée à moins de 0.02 % des naissances, ne constitue qu’un très petit sous-ensemble des nombreuses variantes de la sexuation chromosomique, gonadique et génitale humaine, concernant de l’ordre de 0.4 % à 1.7 % des personnes selon la définition de l’intersexuation retenue et selon les estimations (Blackless et al. 2000 ; Hull 2003 ; Fausto-Sterling 2003 ; Lee et al. 2016).
Les SNP sont les variations entre deux humains les plus fréquentes, et de loin les plus nombreuses en termes de nombre de sites de variation génétique identifiés. Les CNV sont également des variations fréquentes, beaucoup moins nombreuses que les SNP mais couvrant un plus gros pourcentage du génome en nombre de bases, car un seul site de variation de type CNV peut couvrir à lui seul plusieurs centaines, milliers ou même dizaines de milliers de bases, alors qu’un SNP ne touche par définition qu’une seule base.
Une estimation communément admise depuis le début des années 2000 est qu’en moyenne, l’ADN de deux humains de même sexe pris au hasard diffère d’environ 0.1 % en termes de SNP (nombre de différences d’une seule base entre eux / nombre de bases dans un génome haploïde). En particulier, les comparaisons faites en 2007 et en 2010 entre le génome du généticien Craig Venter et un génome de référence, ainsi qu’entre ses deux génomes haploïdes, ont donné à chaque fois 0.1 % de différence (Levy et al. 2007 ; Pang et al. 2010). L’étude de 2007 a estimé la différence en termes de variantes structurelles à 0.4% (exprimée en nombre de bases différentes rapporté au nombre total de bases), mais celle de 2010 faite à l’aide de nouvelles techniques l’a estimée à 1.5%. Les variantes structurelles étant plus complexes à étudier et explorées depuis moins longtemps que les SNP, leur contribution à la variabilité génétique fait l’objet d’estimations plus variables.
Redon et al. (2006) a fait date dans l’exploration des variantes structurelles. Les auteur·es ont recherché chez 270 personnes d’origine européenne, africaine ou est-asiatique les CNV longs de 1 kb ou plus, et estimé qu’ils couvraient au total 360 Mb, soit 12 % du génome. Sur la base de cette étude, il a été estimé qu’ils créaient une différence moyenne de 0.4% entre les génomes de deux personnes (Rotimi & Jorde 2010). L’étude de Conrad et al. (2010) étendue à 450 personnes a permis de valider 8 599 CNV de 500b ou plus, et d’observer qu’ils concernaient 5.5% des gènes codants et 12.5% de tous les gènes. En comparant deux à deux les génomes de 41 personnes de leur échantillon, les auteur·es ont trouvé que ces CNV créaient en moyenne 0.78 % de différence entre elles. Comme le soulignent Zarrei et al. (2015), cette étude est limitée par son manque de prise en compte des petits CNV et par l’utilisation d’un échantillon de populations assez restreint.
En 2008, le projet international « 1000 Genomes » a été lancé pour améliorer la connaissance de la variabilité génétique humaine en constituant un référentiel des variations existantes plus vaste et moins eurocentré que ceux alors disponibles. Les génomes de 2504 personnes originaires de vingt-six populations de cinq régions du monde (661 d’Afrique, 504 d’Asie de l’Est, 503 d’Europe, 489 du sous-continent indien et 347 d’Amérique) ont finalement été séquencés et analysés dans ce cadre.
A partir de cette analyse, les chercheur·es ont estimé que le génome d’un être humain présentait « typiquement 4.1 millions à 5.0 millions de différences avec le génome humain de référence », dont 99.9% de SNP et d’indels courts (1 à 50 bases), mais également des variantes structurelles plus conséquentes affectant plusieurs millions de bases (The 1000 Genomes Project Consortium 2015). Le nombre médian de différences en termes de SNP au niveau des autosomes allait de 3.53 millions pour le groupe originaire d’Europe à 4.31 millions pour le groupe d’origine africaine – ce qui correspond d’après mon calcul à environ 0.12% à 0.15% de différence en nombre de bases rapporté à la longueur des autosomes.
Dans un article issu des mêmes travaux mais focalisé sur les variantes structurelles (Sudmant et al. 2015), les auteurs disent estimer la quantité médiane de variantes structurelles portées par un individu à 18.4 Mb par génome diploïde – soit d’après mon calcul environ 0.3% de différence par rapport au génome de référence, rapportée à la longueur d’un génome diploïde (environ 2 x 3100 Mb). Comme le soulignent Zarrei et al. (2015), la force de cette étude est de couvrir un large échantillon de populations diverses, mais sa portée est limitée par la capacité réduite de la technique utilisée à détecter les CNV de grande taille.
A la lumière de ma revue de la littérature scientifique disponible, il apparaît que l’idée commune selon laquelle il existe en moyenne entre deux humains de même sexe 0.1% de différence en termes de SNP et 0.4% de différences structurelles est mal étayée, et il paraît raisonnable de supposer que ces deux pourcentages sont légèrement sous-estimés. Des projets sont toujours en cours dans plusieurs pays afin d’améliorer la documentation de la variabilité génétique humaine par l’analyse des génomes d’un grand nombre d’individus.
Signification fonctionnelle d’un pourcentage de différence entre génomes humains
Si l’estimation de la différence moyenne entre les génomes de deux humains pris au hasard est fréquemment mobilisée dans le cadre de la vulgarisation scientifique ou à des fins politiques, elle n’est en revanche pas un élément d’étude en soi, car l’exprimer par un chiffre unique revient à agréger des variations dont l’origine, la nature et les conséquences fonctionnelles sont très différentes. A fortiori, calculer « la » différence génétique entre femmes et hommes n’a pas d’intérêt, et n’est d’ailleurs l’objet d’aucun article scientifique. Tout au plus fait-elle (rarement) l’objet de considérations oiseuses en marge d’une publication [3].
En particulier, on ne peut déduire de l’ampleur d’une différence dans l’ADN (que celle-ci soit exprimée en nombre bases ou en nombre de gènes différents) ni l’ampleur de la différence induite en termes d’ARN, ni celle de la différence induite en termes de protéines. A fortiori, l’ampleur de l’écart en termes de phénotype (différences anatomiques, physiologiques, de susceptibilité à telle ou telle maladie…) ne peut être déterminée a priori.
Une différence d’ADN importante en nombre de bases peut n’avoir strictement aucune conséquence si elle affecte une partie non fonctionnelle du génome, et certaines différences au sein de gènes codants, dites synonymes, ne modifient pas la séquence des protéines produites par leur expression. Zarrei et al. (2015) ont même trouvé chez l’humain 107 gènes codants sujets à CNV dont la suppression d’au moins 85% des exons était selon eux « sans conséquences apparentes sur le phénotype ».
A contrario, une différence d’une seule base peut avoir des conséquences très importantes si par exemple elle modifie ou empêche l’expression d’un gène clé. De même, une différence d’un seul gène peut avoir des conséquences considérables : le gène Sry en est un très bon exemple, sa seule présence déclenchant une cascade d’effets notables induits par la présence de testicules fonctionnels. Même une différence de type translocation, c’est-à-dire de zéro gène et zéro base, peut très bien avoir des conséquences notables dès lors que l’expression de la séquence d’ADN déplacée s’en trouve régulée différemment.
De plus, plusieurs différences génomiques peuvent s’annuler au sens où leurs effets se compensent mutuellement, ou parce qu’une différence donnée a pour seul effet d’empêcher une autre de s’exprimer. Ainsi, le gène Xist n’existe pas sur le chromosome Y et ne s’exprime pas dans les cellules XY, mais il a pour effet d’empêcher un grand nombre de gènes du X de s’exprimer davantage dans les cellules XX que dans les cellules XY : ainsi, ce qui est au départ une différence d’ADN « supplémentaire » entre cellules XX et XY les rend en fait plus semblables au final.
Il est cependant toujours possible de voir Xist sous un autre angle, comme le font deux leaders historiques de la recherche sur les différences biologiques entre rongeurs femelles et mâles et entre femmes et hommes, Margaret McCarthy et Artur Arnold (2011) : outre que l’expression de Xist rend femmes et hommes différents dans la mesure où seules les femmes sont porteuses d’un « mosaïcisme » du chromosome X (qui les protége relativement contre les effets d’anomalies du X à l’origine de maladies, comme cela est avéré de longue date) [4], l’expression de Xist « mobilise une vaste machinerie épigénétique qui n’est pas active dans les cellules XY », ce qui pourrait également avoir des conséquences (à ma connaissance non avérées à ce jour).
Différence génomique entre humains et chimpanzés
L’idée que les génomes de l’humain et du chimpanzé sont semblables à 98.5% est très répandue depuis le début des années 1990, mais elle est fausse. Elle est basée sur des calculs qui ont été indûment extrapolés, et surtout mal interprétés, car ils n’avaient pas pour objectif d’estimer « la » différence génomique entre les deux espèces.
L’idée de 1.5% de différence a d’abord été remise en cause en 2001, lorsque Feng-Chi Chen et Wen-Hsiung Li ont publié le résultat de comparaisons entre le génome humain et ceux du chimpanzé, du gorille et de l’orang-outan. Après avoir sélectionné 53 segments communs à ces espèces et non dupliqués de régions non codantes (« car elles ne sont pas directement soumises à la sélection naturelle et permettent donc de retracer plus précisément l’histoire évolutive » [Chen & Li 2001, ma traduction]), les auteurs ont calculé le nombre moyen de nucléotides différents entre espèces dans ces segments. Entre humain et chimpanzé ils ont trouvé 1.24 % de différence moyenne, soit nettement moins que l’estimation prévalant alors, notamment parce que celle-ci incluait des séquences dupliquées. Ils ont tenu à souligner que leur objectif était d’estimer les divergences dans des régions non dupliquées, et que celles existant dans les séquences répétitives « pourraient être nettement plus grandes » du fait d’un taux de mutation plus élevé et de fréquents indels dans celles-ci.
Dès l’année suivante, après avoir examiné les mêmes échantillons d’ADN que ceux utilisés par des chercheur·es venant de publier une estimation très proche de celle de Chen et Li, Roy Britten publie le résultat d’un calcul incluant non seulement les séquences répétées, mais aussi les indels. Il trouve une différence moyenne de 1.4 % en termes de substitutions de nucléotides (séquences répétées incluses), à laquelle viennent s’ajouter 3.4 % de différence en termes d’indels. Sa conclusion forme le titre de son article publié dans PNAS : « La divergence entre échantillons de séquences d’ADN du chimpanzé et de l’humain est de 5 % en comptant les indels » (Britten 2002).
En 2005, un consortium international publie dans Nature le premier brouillon de séquençage presque complet du génome du chimpanzé, ainsi que le résultat d’une comparaison avec le génome humain (The Chimpanzee Sequencing and Analysis Consortium 2005). Au sein des séquences ayant pu être alignées – ne couvrant en l’occurrence qu’environ 2400 Mb sur plus de 3000 Mb dans un génome haploïde (seuls 13 454 gènes codants humains ont pu être mis en correspondance avec des gènes du chimpanzé) –, l’article rapporte une divergence moyenne de 1.23 % en termes de substitution d’un seul nucléotide, avec un minimum de 0.94 % pour le chromosome X et un maximum de 1.9 % pour le Y. Les auteur·es estiment que les indels génèrent une différence additionnelle d’environ 3 %, répartie en gros à 50/50 : environ 1.5 % de différence correspondent à des bases en plus dans le génome humain, et environ 1.5 % à des bases en plus dans celui du chimpanzé. L’étude permet d’avancer une nouvelle estimation de la différence d’environ 4 % en termes de séquences d’ADN, comme l’annonce alors dans Le Monde le journaliste scientifique Hervé Morin (01/09/2005), mais elle n’inclut pas tout.
En effet, tout d’abord, comme cela est signalé dans les notes supplémentaires en ligne – et également souligné par Varki & Nelson (2007), ou encore Franchini & Pollard (2017) –, la méthode employée pour comparer les deux génomes a ignoré par construction un certain nombre de segments délétés ou dupliqués. Ensuite, outre les différences en termes de séquences, l’article de 2005 rappelle l’existence de différences correspondant à des réarrangements de grande ampleur : l’homologue du chromosome 2 humain correspond à deux chromosomes chez le chimpanzé, et au moins neuf vastes portions d’ADN sont inversées entre les deux génomes (sur les chromosomes 1, 4, 5, 9, 12, 15, 16, 17 et 18). On ignore les effets de ces réarrangements sur l’expression des gènes. Relevant que la fusion des chromosomes 2a et 2b ainsi que les inversions sur le 1 et le 18 sont spécifiques à la lignée humaine, et que les sept autres inversions sont communes au chimpanzé et au bonobo, Varki et Nelson (2007) ont suggéré que cela pourrait être lié à l’émergence de spécificités humaines depuis la séparation entre notre lignée et celle de ces deux espèces.
Un spécialiste de l’évolution du cerveau des primates a résumé la situation en ces termes en 2012 : « la similarité entre la séquence totale d’ADN des humains et celle des chimpanzés n’est pas de 98 % ou 99 %, mais plutôt plus proche de 95 % à 96 % », et les réarrangements sont en outre « si vastes » qu’ils rendent « excessivement simpliste » une telle comparaison unidimensionnelle (Preuss 2012, p. 10711, ma traduction). De plus, les données disponibles à ce jour indiquent que la différence réelle en termes de séquence d’ADN est sans doute supérieure à cette estimation de 4 à 5 % [5].
Quoi qu’il en soit, la taille de cette différence génomique multidimensionnelle ne permet nullement d’inférer une taille similaire de différence phénotypique. Il est clair qu’elle a des conséquences considérables sur les plans anatomique, physiologique ou encore cognitif, incommensurables aux différences existant en moyenne entre deux humains, mais on ne sait pas encore par quels biais (Varki & Altheide 2005 ; Franchini & Pollard 2017). Il est clair également que de petites différences en pourcentage d’ADN peuvent induire des différences biologiques plus que notables. Par exemple, Glazko et al. (2005) se sont demandé comment la différence d’un peu plus de 1 % en termes de substitution de nucléotides se traduisait en termes de structure chimique des protéines produites. Sur la base d’une analyse de 127 protéines codées par des gènes orthologues [6] très similaires entre humain et chimpanzé, leur article rapporte que malgré leur 1 à 2 % de différence seulement, 80 % des protéines codées par ces gènes étaient différentes. Autre exemple : Cheng et al. (2005) se sont demandé comment la différence d’un peu moins de 3 % en termes de duplications de segments de plus de 20 Kb se traduisait en termes de niveau d’expression des gènes concernés par ces duplications. Basé sur la comparaison dans cinq tissus (cerveau, cœur, reins, foie et testicules) de l’expression de 177 gènes dupliqués seulement chez l’humain (88 en entier, 89 en partie) et de 94 gènes dupliqués seulement chez le chimpanzé, leur article rapporte que 56% des premiers et 49% des seconds avaient un niveau d’expression significativement différent entre les deux espèces dans au moins l’un des cinq tissus.
PARTIE 2 – BREVE HISTOIRE DE L’ARGUMENT DE CLAUDINE JUNIEN
Début des années 2000 : Mattei, Ginger et compagnie
Au début des années 2000, la génomique comparative en est encore à ses balbutiements, le nombre de gènes humains reste flou, l’ADN non codant est volontiers appelé « ADN poubelle » car on le pense essentiellement non fonctionnel, et on ignore encore largement la contribution des variations structurelles à la variabilité génétique. Des « différences génétiques » moyennes entre deux hommes et entre humain et chimpanzé ont été calculées en nombre de bases, ne tenant compte que des SNP pour la première et des substitutions de nucléotides au sein d’échantillons de séquences alignées pour la seconde. Les estimations de ces différences qui prévalent alors sont de 0.1 % pour celle entre deux hommes, et de 1.5 % à 1.6 % pour celle entre humain et chimpanzé.
Ces estimations sont reprises dans un ouvrage collectif sur le génome humain publié en 2001 par le Conseil de l’Europe, dirigé par un autre professeur de génétique médicale et membre de l’Académie nationale de médecine : Jean-François Mattei (également député à l’origine des lois de bioéthique de 1994). L’estimation de la différence entre humain et chimpanzé y est comparée à une estimation grossière de la distance génétique entre femmes et hommes : sachant qu’en nombre de bases, le chromosome X correspond à environ 5 % de la longueur du génome humain, l’auteur en déduit qu’étant donné que les femmes ont deux chromosomes X et les hommes un seul, cela « pourrait signifier que les hommes et les femmes sont différents à 5%, c’est-à-dire davantage que les humains et les chimpanzés du même sexe » [7].
Ce raisonnement à l’emporte-pièce incorpore une grossière erreur logique, car la longueur totale du génome humain utilisée pour arriver à 5 %, à savoir 3 milliards de bases (5% = environ 150 Mb sur le chromosome X / environ 3000 Mb en tout), est celle d’un génome haploïde. Si on prend en compte la seconde copie du X, il faut incorporer aussi au dénominateur la seconde copie du X et celles de tous les autosomes. Les génomes des femmes et des hommes ne diffèrent alors déjà pas de 5 % en nombre de bases, mais de 2.5 % (il est bien indiqué ailleurs dans l’ouvrage que le génome diploïde est long d’environ 6 milliards de bases). En outre, la comparaison entre ces deux « différences génétiques » est pour le moins spécieuse, car le pourcentage de différence entre humain et chimpanzé mobilisé ici correspond à des différences de code génétique au sein de séquences homologues, alors que celui entre femmes et hommes correspond à des différences de quantité de séquences de code génétique identique. L’auteur de la comparaison est d’autant plus malhonnête – ou aveuglé par ses préjugés sexistes – que le mécanisme d’inactivation d’un X sur deux dans les cellules XX est connu de longue date, et qu’il ne peut ignorer que cela empêche la plupart des gènes du second X de s’exprimer.
Le même calcul foireux et la même comparaison sont à l’époque diffusés et instrumentalisés par le psychothérapeute Serge Ginger, important promoteur en France des mythes savants imprégnés du sexisme le plus crasse concernant les différences femmes-hommes dont il se prétendait expert (et était relayé comme tel dans les médias) [8]. Dans une conférence intitulée « Cerveau féminin / Cerveau masculin » donnée maintes fois à partir de 2001 et mise en ligne en 2003, égrainant une liste ébouriffante de différences femmes-hommes supposées naturelles, présumée vulgariser des acquis des neurosciences mais en réalité tirée de sa lecture d’une littérature de (pseudo)vulgarisation allant de John Gray à Doreen Kimura en passant par Boris Cyrulnik ou encore Jean-Didier Vincent [9], Ginger affirme ceci : « En fait, nous appartenons à deux “espèces” différentes ! À cette époque où l’on vient d’achever les premières phases de décryptage du génome humain, vous savez peut-être qu’on a pu montrer que l’homme et le singe possèdent un patrimoine génétique de base, commun à 98,4 % ; ce qui laisse 1,6 % de différence seulement… contre environ 5 % de différence génétique entre l’homme et la femme. Ainsi, un homme mâle est physiologiquement plus proche d’un singe mâle que d’une femme ! ».
L’argument est si frappant qu’il est notamment repris, accompagné des élucubrations de Ginger sur les différences cognitives et comportementales entre femmes et hommes, dans un guide de développement personnel écrit par deux gestalt-thérapeutes sexologues, dont l’ex-président de la Société Française de Sexologie Clinique (Gellman et Higy-Lang 2003, p.61) – ce qui n’a pas gêné l’auteur de la préface Philippe Brenot, sexologue et à l’occasion également expert médiatique de différences entre femmes et hommes dans le domaine de la sexualité. On retrouve encore cet argument dans un livre récent censé vulgariser des acquis des neurosciences et écrit par deux professeur·es à HEC qui se piquent de donner des conférences en neurosciences [10] mais citent en référence Serge Ginger (!) (Badoc et Bayle-Tourtoulou 2016, p. 114). Outre que les propos de Serge Ginger relevaient déjà du grand n’importe quoi en 2001-2003, de l’eau a pourtant coulé sous les ponts depuis, que ce soit en neurosciences ou en génétique. En particulier, de nouvelles données ont été publiées concernant le génome du chimpanzé comme on l’a vu plus haut, mais aussi concernant les gènes échappant au processus d’inactivation du X chez l’être humain, comme on va le voir maintenant.
2005 : Carrel et Willard publient des données à prendre avec des pincettes
En 1999, les travaux issus du post-doctorat de Laura Carrel et de deux autres chercheuses au sein du laboratoire de Huntington Willard ont permis à Carrel et al. (1999) de publier dans PNAS une étude au terme de laquelle elles et il ont estimé (par extrapolation) que 15 % des gènes du X humain échappaient à l’inactivation d’un X sur deux dans les cellules XX [11]. En 2005, le premier séquençage quasi-complet (99.3%) du chromosome X humain auquel tous deux ont participé leur permet d’affiner cette estimation, et – Ô miracle – de retomber pile sur 15 %.
Carrel et Willard (2005) sont cette fois seuls co-auteurs de l’étude. Leur article est publié dans Nature en même temps que celui synthétisant les résultats du séquençage du chromosome X (Ross et al. 2005), qui a permis de localiser 1098 gènes sur le chromosome X (699 déjà répertoriés, 132 nouveaux gènes codants, 166 nouveaux gènes non codants et 101 gènes putatifs), ainsi que 700 pseudogènes. Carrel et Willard ont de leur côté étudié l’expression de 624 gènes réels ou putatifs (dont 471 déjà répertoriés) sur des chromosomes X inactivés (Xi).
Ce nombre réduit est notamment dû au fait que ces gènes étaient les seuls dont l’expression était détectable dans les cellules utilisées pour leur étude, à savoir d’une part des fibroblastes humains prélevés dans la peau de 40 femmes, et d’autre part des cultures de neuf lignées cellulaires hybrides souris-humain issues de neuf femmes, également de fibroblastes de la peau [12]. L’expression des gènes des Xi a été estimée à l’aide de méthodes complexes, dont une méthode originale mise au point par Carrel et Willard pour les fibroblastes humains.
Seuls 94 gènes ont été testés dans les fibroblastes humains [13]. L’article rapporte que 20 % n’étaient inactivés que dans « certains » échantillons et que 15 % « échappaient à l’inactivation » dans tous. L’article souligne que « la plupart » des gènes échappant à l’inactivation ne s’exprimaient toutefois que « partiellement », sans plus de précision. On sait juste (Table S4) qu’un gène du Xi a été jugé exprimé dans un échantillon de fibroblaste humain si son niveau d’expression était égal à au moins 6 % de son niveau d’expression sur le X activé (Xa), et la Figure 2 indique que le niveau d’expression sur le Xi était souvent faible, rarement supérieur à la moitié de celui du Xa.
Par ailleurs, 624 gènes réels ou putatifs ont été testés dans les lignées hybrides, la Table S3 en ligne permettant de voir qu’ils incluaient 12 gènes des régions PAR ainsi que le gène Xist lui-même (échappant évidemment à l’inactivation). Pour ce second test, un gène du Xi a été jugé exprimé si son niveau d’expression était égal à au moins « 10 à 15% » de son niveau d’expression sur Xa (Note s2). La figure ci-après récapitule les résultats synthétisés dans la Table S3 en ligne.
Le texte de l’article présente la synthèse concernant « les 401 gènes pour lesquels les résultats étaient complètement concordants dans tous les hybrides », ce qui est bien égal à 325 + 76, la somme des lignes « Aucun » et « Tous » du tableau ci-dessus. Selon l’article, 74 ont échappé à l’inactivation dans tous les hybrides (76 selon la Table S3), ce qui après exclusion des gènes de PAR1 correspond à « au moins 16 % » des gènes testés, un pourcentage « globalement identique à l’estimation tirée des études faites sur les cellules humaines ». Il ressort pourtant de la Table S3 que seulement 12 % des gènes testés ont systématiquement échappé à l’inactivation, et après exclusion des gènes de PAR1 on arrive à moins de 11 % (=66/(623-10)). Leur conclusion selon laquelle environ 15 % de gènes « échappent à l’inactivation », et environ 10 % supplémentaires y échappent « dans un sous-ensemble significatif des chromosomes X testés » ne peut être retrouvée qu’en faisant les regroupements arbitraires montrés dans le tableau ci-dessus, c’est-à-dire en classant comme « échappant » les gènes ayant échappé dans au moins 7 lignées hybrides sur 9 (selon Cotton et al. 2013 p. 4, c’est effectivement ce critère que Carrel et Willard ont utilisé), et comme « inactivés » ceux inactivés dans au moins 7 lignées sur 9.
Pour ce qui est de la cohérence entre les tests faits sur les fibroblastes humains et ceux faits sur les hybrides, aucune donnée détaillée n’est disponible. Carrel et Willard se contentent d’indiquer que les résultats des deux tests étaient en accord « dans la plupart des cas », signalant un seul exemple de discordance : sur les 20 gènes échappant à l’inactivation dans toutes les lignées hybrides et dont l’expression a aussi été testée dans les fibroblastes humains, seuls 12 échappaient à l’inactivation dans tous les échantillons humains [14]. Ce sont donc sans doute nettement moins que 15 % des gènes qui échappent systématiquement à l’inactivation chez les femmes (tout au moins dans les fibroblastes de la peau), mais Carrel et Willard préfèrent souligner que cela suggère que ce sont peut-être encore plus que 10 % des gènes du X qui échappent ou non à l’inactivation selon les femmes.
Pour reprendre les mots du commentaire enthousiaste de cette étude fait par la généticienne Mary Lyon (2005), découvreuse de l’inactivation du X en 1961 et ayant suggéré dès 1962 que certains gènes pouvaient y échapper, « si ces résultats obtenus sur des cultures cellulaires reflètent la situation in vivo », cela pourrait avoir des implications importantes. Mais comme elle le souligne, le processus d’inactivation du X est « moins stable que la normale » dans les cultures de cellules hybrides, « au sens où les traitements expérimentaux peuvent réactiver certains gènes du X » (ma traduction). Elle signale aussi que chez certains marsupiaux au moins, les gènes du Xi sont inactivés dans les tissus du corps mais réactivés dans les fibroblastes de la peau. Par conséquent, selon elle « il semble possible » que chez l’humain aussi, « bien que la majorité des gènes restent inactivés de manière stable dans les fibroblastes de la peau, certains puissent être réactivés tout en restant réduits au silence in vivo ». Par ailleurs, elle ajoute que « si des gènes échappent réellement in vivo de manière variable à l’inactivation du X, il sera intéressant de savoir s’ils se comportent de la même façon dans toutes les cellules d’une femme donnée. Il semble probable que ce n’est pas le cas ».
Bien-sûr, la médiatisation de l’étude ignorera les incohérences que j’ai relevées ci-dessus et ne s’embarrassera pas de ses limitations intrinsèques telles que celles soulignées par Mary Lyon.
Publicité/communication des résultats de Carrel et Willard
Le 16 mars 2005, veille de la parution du nouveau numéro de Nature, l’employeur de Huntington Willard émet un communiqué de presse (Duke University Medical Center 2005, extraits trad. par moi ci-après). L’annonce est relayée par le groupe de presse qui possède Nature dans le cadre de sa sélection-synthèse périodique envoyée aux journalistes, ainsi que par le fil d’actualité Eurekalert! géré par l’American Association for the Advancement of Science, une autre source de communiqués de presse communément utilisée par les journalistes scientifiques.
Histoire de camper Willard en découvreur, il y est expliqué qu’alors que les scientifiques avaient initialement supposé que le processus d’inactivation du X empêchait complètement les gènes du X inactivé de s’exprimer, « Willard et d’autres » avaient remis en cause cette hypothèse en apportant, à la fin des années 1980, les premières preuves du contraire. S’il est exact que Willard travaille sur le sujet depuis cette époque, c’est en réalité en 1979 et en 1984 que d’autres que lui ont publié les premières observations d’échappement de gènes du X à l’inactivation (Shapiro et al. 1979 ; Goodfellow et al. 1984), en l’occurrence le gène STS, codant pour une enzyme impliquée dans la synthèse des hormones stéroïdiennes, et le gène CD99 (ex MIC2) appartenant à la région PAR1.
Après avoir prétendu à tort que « le niveau d’activité de 471 gènes » a été mesuré par les chercheurs dans des lignées cellulaires issues de 40 femmes, le communiqué indique qu’ils ont « trouvé que globalement, environ 15 % des gènes du second chromosome X échappent dans une certaine mesure à l’inactivation ». De plus, « chez certaines femmes mais pas chez d’autres, 10 % supplémentaires des gènes du X présentent des profils d’inactivation variables et des niveaux d’activité différents selon les chromosomes X “inactifs”. […] Selon Huntington Willard, […], les résultats de cette étude pourraient avoir d’importantes implications pour la compréhension des différences entre femmes ainsi qu’entre hommes et femmes, tant en matière de santé que de maladie, et ils ouvrent également une nouvelle perspective sur les fondements des différences bien établies entre les sexes ».
En effet, nous explique-t-on, outre que seules les femmes sont constituées d’une « mosaïque de deux types cellulaires » (puisque ce n’est pas toujours le même X qui est inactivé) et que le chromosome Y « confère également aux hommes au moins plusieurs dizaines de gènes exprimés que les femmes n’ont pas », l’incomplétude de l’inactivation du X « mise en évidence » par cette nouvelle étude « signifie qu’au moins 15 % des gènes du X, ainsi que leurs produits protéiques, sont présents à des niveaux caractéristiquement plus élevés chez les femmes que chez les hommes [rem : niveaux non rapportés par Carrel & Willard], et souvent variables chez elles. De plus, les résultats montrent qu’au moins 10 % de gènes supplémentaires sont exprimés à des niveaux variables selon les femmes, tandis que tous les hommes expriment une seule copie de ces gènes. “Nous savons maintenant que 25 % du chromosome X – 200 à 300 gènes – peut être exprimé d’une façon spécifique à l’un des deux sexes“, a déclaré Willard. “En substance, il n’y a pas un génome humain, mais deux : le masculin et le féminin. Cette différence génomique devrait être reconnue en tant que potentiel facteur explicatif des caractéristiques spécifiques à un sexe de maladies complexes, ainsi que des différences normales entre les sexes“».
A la fin du communiqué, Willard indique qu’il reste notamment à déterminer « si le profil d’inactivation du X varie selon les tissus ou au fil du temps ». Pourtant, et alors que l’article indique en outre que pour la plupart des gènes échappant à l’inactivation, le niveau d’expression était loin de s’en trouver doublé, la généticienne éditrice pour Nature des études de génétique annonce en fanfare dans les pages « News and views » du journal que l’étude a montré qu’environ 15 % des gènes du X échappent « de façon permanente à l’inactivation, ce qui signifie que leur niveau d’expression est le double chez les femmes » (Gunter 2005).
L’employeur de Laura Carrel émet également un communiqué de presse qui sera relayé par ScienceDaily (Penn State 03/2005), dont le contenu est plus correct. En particulier, Carrel y indique que le premier des deux tests a porté sur des cellules de peau humaine et seulement 94 gènes, que les résultats du second, ayant porté sur 624 gènes, sont tirés d’« expériences réalisées sur des cultures cellulaires » (ce qui laisse au moins entrevoir que l’extrapolation aux cellules in vivo n’est pas évidente), et que « les implications cliniques restent entièrement à explorer ». Les résultats « pourraient avoir des implications pour la prise en charge des personnes porteuses d’anomalies du chromosome X », dit Carrel, et « pourraient expliquer certaines des différences entre hommes et femmes qui ne peuvent être attribuées aux hormones sexuelles ». Moins sexy que l’autre communiqué de presse, celui-ci ne contient pas l’idée de Willard selon laquelle il faudrait désormais considérer qu’il existe deux génomes humains, un féminin et un masculin, mais introduit pour sa part l’idée que l’étude conforte « les hommes qui pensent que les femmes sont complexes » : ils ont manifestement raison, « au moins en termes de gènes ».
Relais fantasque de l’étude de Carrel et Willard dans les médias anglo-saxons
Steve Connor, journaliste scientifique multi-primé du journal britannique The Independent, est l’un des premiers [15] à dégainer pour faire du résumé de l’étude un scoop à sensation. Selon lui, on vient de découvrir que la « guerre des sexes », qui prend racine dans « 300 millions d’années de lutte entre les chromosomes X et Y », a rendu « le génome féminin très différent de celui des hommes » (Connor 17/03/2005, ma traduction). Lorsqu’il est chez les femmes, le chromosome X « se comporte si différemment de lorsqu’il est chez les hommes qu’en fait, cela a abouti à la formation d’un autre génome humain » : « […] “En substance, il n’y a donc pas un génome humain, mais deux : le masculin et le féminin“, a déclaré le Professeur Willard ».
Alors que Laura Carrel indique prudemment que leur étude « pourrait expliquer certaines des différences entre hommes et femmes qui ne sont pas attribuables aux hormones sexuelles », Steve Connor reformule abruptement : « Autrement dit, les différences physiques et émotionnelles entre hommes et femmes pourraient être plus profondes que celles qui sont causées simplement par les hormones. L’étude a ainsi montré, par exemple, que 15% des gènes qui sont actifs sur le chromosome X “inactif” sont présents [sic] à un plus haut niveau chez les femmes que chez les hommes ».
Pour Robert Lee Hotz, journaliste scientifique du Los Angeles Times également multi-primé (et finaliste en 1986 du prix Pulitzer pour une enquête sur les impacts de l’ingénierie génétique), dans cette étude publiée en même temps que ce qu’il qualifie de premier « décodage complet » du chromosome X, « les scientifiques ont trouvé des éléments de preuve génétique de ce que certains hommes soupçonnent depuis longtemps : il est risqué de faire des suppositions sur les femmes » (Hotz 17/03/2005, ma traduction). En effet, « le comportement de nombreux gènes qui sont au cœur de l’identité sexuelle peut rendre les femmes presque aussi différentes les unes des autres qu’elles le sont des hommes ».
« L’analyse a également montré que les différences obsessionnellement débattues entre hommes et femmes étaient, au moins sur le plan génétique, encore plus grandes qu’on ne le pensait. Jusqu’à 300 des gènes du chromosome X peuvent être activés différemment chez les femmes […] Cette variabilité génétique nouvellement découverte chez les femmes pourrait contribuer à expliquer les différences entre les sexes dans la réaction aux médicaments et la vulnérabilité accrue des femmes à certaines maladies, ont déclaré les experts. […] Tout compris, la différence entre hommes et femmes pourrait s’élever à 2 % de l’ensemble de leur patrimoine génétique [16], soit plus que le fossé héréditaire séparant l’humanité de son parent le plus proche, le chimpanzé. “En substance, a déclaré Willard, il n’y a pas un génome humain mais deux : le masculin et le féminin. ».
Maureen Dowd, éditorialiste au New York Times connue pour sa sensibilité « féministe » et journaliste reconnue (lauréate du prix Pulitzer pour ses articles sur l’affaire Bill Clinton/Monica Lewinsky), propose une interprétation assez différente, mais partage avec Connor la personnification du substrat génétique [17], ainsi que l’idée déjà exprimée par Connor et Hotz que ces recherches révèlent une différence profonde et d’ordre psycho-comportemental entre hommes et femmes. Elle retient notamment ces propos de l’interview qu’elle a faite de Willard : « les hommes et les femmes sont encore plus éloignés les uns des autres qu’on ne le pensait ; ce n’est pas soit Mars, soit Vénus, c’est soit Mars, soit Vénus, Pluton, Jupiter ou on ne sait quelles autres planètes » (Dowd 20/03/2005, ma traduction). « Nous autres, pauvres hommes, n’avons que 45 chromosomes pour faire notre job car notre 46ème est le pathétique Y, pourvu de seulement quelques gènes qui opèrent entre le genou et la ceinture », poursuit Willard, alors que les femmes ont un 46ème chromosome « entier » et qui est plus actif qu’on ne le pensait. C’est pourquoi les hommes sont « les créatures simples qu’ils ont l’air d’être », explique Dowd, alors que les femmes sont « génétiquement plus complexes ». Pour elle, cela signifie que les hommes ont raison de dire que les femmes sont « impénétrables, changeantes, retorses, singulières », en somme « une espèce différente ».
Voilà qui « pourrait aider à expliquer pourquoi le comportement et d’autres caractéristiques des hommes et des femmes sont si différents : ils sont peut-être câblés en dur dans le cerveau, en plus de leurs causes hormonales ou culturelles ». Dowd se demande si Lawrence Summers n’a finalement pas raison (le président de Harvard vient alors de faire scandale en déclarant que le faible pourcentage de femmes dans les postes de haut niveau en science et ingénierie pourrait être dû à leurs moindres aptitudes dans ces domaines), et conclut en suggérant que « cette découverte concernant l’expression supérieure des gènes chez les femmes pourrait peut-être répondre à la vieille question de savoir pourquoi les hommes ont du mal à s’exprimer : parce que leurs gènes ont du mal à le faire ».
Pour Fred Guterl aussi, le responsable des informations scientifiques et techniques de Newsweek, l’étude est du pain béni : ce qui est ici en question, c’est rien moins que « la vérité sur le genre » (Guterl 28/03/2005, ma traduction). Significativement, lui aussi relie cette étude à la question des différences cognitives et comportementales entre femmes et hommes, évoquant pour introduire son papier le cas édifiant d’une petite fille que des parents ont tenté en vain d’élever de manière non sexiste, mais qui joue à la poupée avec ses camions (« après tout, les enfants sont bien plus aptes que les adultes à exprimer leur nature fondamentale »), mais aussi l’affaire Larry Summers (« Summers est peut-être coupable de dire quelque-chose de socialement incorrect, mais a-t-il tort scientifiquement parlant ? La biologie ne joue-t-elle aucun rôle notable dans la détermination des talents et des comportements des hommes et des femmes ? »).
Selon Guterl, cette étude publiée il y a une semaine dans Nature, venant s’ajouter à toutes celles qui ces dernières années, ont montré que « le fossé biologique entre hommes et femmes est plus large qu’on ne le pensait », a observé qu’ « environ 20 % » des gènes du second chromosome X restent actifs chez les femmes, « soit environ 200 gènes ». Il s’agit de « l’une des observations les plus intrigantes », et elle concerne leur différence génétique : « [l’étude] évalue cette différence à environ 1 %. Sachant que le bagage génétique des chimpanzés ne diffère de celui des humains que de 1.5 %, c’est significatif. “On pourrait dire qu’il y a deux génomes humains, un pour les hommes et l’autre pour les femmes“, affirme Huntington Willard ».
Un aspect original du récit de Fred Guterl, concernant les effets présumés des hormones dites « sexuelles », est à souligner. Bien qu’il le fasse très maladroitement, il a le mérite de pointer du doigt le fait que les recherches menées depuis des décennies laissent peu de doute : si d’aventure la testostérone produite in utero par les testicules des garçons s’avérait induire une masculinisation/déféminisation de leur cerveau se traduisant ensuite en termes cognitifs et comportementaux, cet effet présumé de la testostérone dans notre espèce ne saurait être d’une ampleur notable (voir à ce sujet Jordan-Young 2016). C’est pourquoi les scientifiques qui travaillent sur la question des bases biologiques de certaines différences cognitives ou comportementales observées entre femmes et hommes s’intéressent de plus en plus à d’autres hypothèses, dont celle d’un effet direct de l’expression différente de certains gènes dans les cellules du cerveau.
La façon dont Guterl le formule, significative, vaut la peine d’être citée : « Les scientifiques ont découvert que bien que les hormones causent des ravages sur presque tous les aspects de la physiologie des adolescents, elles n’ont presque aucun effet sur le développement du cerveau. Les études faites sur des triplés où des filles sont prises en sandwich entre deux frères dans le ventre de leur mère montrent ainsi que même si ces filles acquièrent certains traits masculins, en raison d’une dose de testostérone plus forte que la normale, leur cerveau n’est pas affecté. […] Qu’est-ce qui fait, donc, que les cerveaux des femmes sont différents de ceux des hommes ? » Le journaliste croit savoir que « les variations génétiques », au contraire des hormones, « ont un énorme impact sur le cerveau », et c’est pourquoi cette étude lui paraît prometteuse pour expliquer ce qui est selon lui bien acquis : hommes et femmes utilisent des stratégies de navigation spatiale différentes, ils n’utilisent pas les mêmes circuits cérébraux lorsqu’ils font rimer des mots, les femmes ont 15 à 20 % de matière grise que les hommes, « c’est-à-dire de neurones ordinaires », et leur matière blanche est concentrée entre les hémisphères de sorte que cela les aide peut-être dans les tâches langagières, sans compter qu’aux examens d’entrée à l’université, « les femmes obtiennent systématiquement des résultats inférieurs à ceux des hommes en mathématiques ».
Relais plus factuel dans les médias français, faux et trompeur par Peggy Sastre
L’étude fait moins sensation en France, où l’on n’hésite pourtant pas à relayer et instrumentaliser tout ce qui peut concourir à naturaliser les différences entre femmes et hommes. Peut-être est-ce parce qu’on est ici de longue date biberonné à la théorie selon laquelle « les hormones » font toute la différence entre eux, et peut-être aussi parce que la méfiance vis-à-vis du « tout génétique » y est plus développée qu’aux Etats-Unis ou en Grande Bretagne.
Quoi qu’avec les types de raccourcis et défauts habituels (ce résultat à prendre avec des pincettes est présenté comme acquis, l’in vitro est extrapolé à l’in vivo, des cellules de la peau de moins de 50 femmes états-uniennes sont présumées révéler ce qui se passe dans toutes les cellules du corps de toutes les femmes, etc), ainsi qu’avec diverses erreurs factuelles [18] et quelques glissements sémantiques signifiants [19], l’étude est relayée d’une manière qui reste plus factuelle. En particulier, on n’y retrouve ni l’idée des « deux génomes », ni le calcul de « la » différence génétique entre femmes et hommes, ni la comparaison avec celle nous séparant du chimpanzé (do O’Gomes 17/03/2005 ; Nau 19/03/2005 ; RTFlash 24/03/2005 ; Laroque 30/03/2005 ; Gilgenkrantz 2005).
La future « journaliste scientifique » Peggy Sastre se jette en revanche avec gourmandise sur l’étude de Carrel et Willard, dans un billet publié (anonymement) sur un site bien particulier : il relaie de manière ciblée les résumés d’articles scientifiques susceptibles d’étayer la théorie des inégalités cognitives naturelles entre groupes sociaux et ethniques à laquelle elle adhère [20]. Ou disons plutôt qu’elle se jette sur le communiqué de presse émis par le centre de recherche qui emploie Willard, car elle en reprend l’affirmation que l’expression de 471 gènes a été étudiée chez 40 femmes, ce qu’on découvre vite faux lorsqu’on se donne la peine de lire l’article. Sous un titre aux points de suspensions évocateurs – « Deux sexes, deux génomes… », le billet insiste : « Il n’y a pas un génome humain, mais bien deux : le génome mâle et le génome femelle ». Environ 25 % du chromosome X, soit « 200 à 300 gènes », s’expriment de telle sorte que cela diffère « radicalement de la situation des hommes » et que cela crée une « hétérogénéité féminine […] totalement absente chez l’homme […] Les différences sexuelles sont donc inscrites au cœur du génome, dès la conception, pour s’exprimer ensuite tout au long du développement de l’individu. »
2006 : mise en question de la portée des résultats de Carrel et Willard
L’année suivante, une étude mettant sérieusement en question la portée de celle de Carrel et Willard est publiée par Talebizadeh et al. (2006). Afin de savoir dans quelle mesure les résultats de Carrel et Willard obtenus sur les cultures hybrides reflètent ce qui se passe in vivo et se traduisent par une différence entre femmes et hommes, elle et ils ont analysé les données disponibles dans un base partagée de mesures de l’expression de 299 gènes du X et 7795 gènes d’autosomes “in vivo” (par quantification de l’ARN dans un prélèvement) dans onze types de tissus humains (cerveau, rein, foie, poumon, colon, muscle squelettique…). Ces tissus provenaient d’un petit nombre de sujets – dix au maximum pour certains, dont seulement quatre femmes et trois hommes pour le tissu cérébral –, avec souvent une différence importante d’âge moyen entre femmes et hommes. Pour chaque gène et chaque type de tissu, les auteur·es ont calculé le niveau d’expression moyen dans les tissus féminins et dans les masculins, et calculé le ratio F/M correspondant.
En moyenne sur les onze tissus, dans un tissu donné, 5.1 % des gènes du X étaient dans le premier quartile (ratio moyen > 1.5, i.e. gènes nettement « surexprimés » dans les tissus féminins), mais 7.4 % l’étaient au contraire dans le dernier quartile (< 0.7, i.e. « sous-exprimés »). Cela ne pointe déjà pas vers l’idée d’une « surexpression » caractéristique de nombreux gènes du X chez les femmes. Malgré la grande hétérogénéité et la petite taille de l’échantillon, le ratio moyen par tissu de l’expression des 299 gènes était très proche de 1, le minimum étant de 0.97 pour l’iléon (une partie de l’intestin grèle) et le maximum de 1.09 pour le poumon et l’estomac. De plus, la moyenne des ratios F/M ne différait pas significativement entre gènes du X et gènes des autosomes.
En outre, les auteur.es expliquent qu’elle et ils s’attendaient à ce que pour la majorité des gènes dont Carrel et Willard avaient rapporté qu’ils échappaient à l’inactivation, le niveau d’expression soit dans le premier quartile, i.e. supérieur d’au moins 50 % dans les tissus féminins, mais que cela na pas été le cas : « aucune cohérence n’a été détectée entre les résultats générés pour les 217 gènes du X étudiés dans le système hybride de cellules somatiques [de Carrel et Willard] et le ratio F/H calculé à partir de notre analyse ». En particulier, l’article rapporte l’absence de différence statistiquement significative entre tissus féminins et masculins pour le gène SYBL1 de PAR2, qui (étonnamment) était inactivé dans 8/9 lignées de Carrel et Willard.
Par ailleurs, bien que pour certains gènes une tendance ait été détectée, il n’y avait pas de corrélation statistiquement significative entre les niveaux d’expressions dans les différents tissus. Cela pourrait être dû à la trop petite taille de l’échantillon, mais une table supplémentaire en ligne permet de se faire une idée de l’énorme variabilité du ratio F/H selon les tissus, ainsi que de l’impossibilité d’établir un lien entre ces données et celles de Carrel et Willard. Pour n’en citer que la première ligne, on voit ainsi qu’un gène échappant selon ces derniers systématiquement à l’inactivation a pourtant ici un ratio F/H d’expression proche de 1 dans deux tissus, inférieur à 1 dans huit tissus, dont seulement 0.7 dans le cerveau (soit une expression en moyenne inférieure de 30 % dans les tissus féminins), mais égal à 3.4 dans l’estomac (soit une expression supérieure de 240 % !).
Cette table permet aussi de se faire une idée des incohérences entre les deux tests faits par Carrel et Willard, ainsi que du faible surcroît d’expression de certains des gènes « échappant à l’inactivation » mesuré par eux dans les fibroblastes humains. Ainsi, on voit un gène « échappé » dans 9/9 lignées hybrides ayant pourtant un niveau d’expression estimé par eux sur Xi dans les cellules humaines égal à seulement 0.5 % de celui du Xa, et à l’inverse un gène échappé dans 0/9 lignées hybrides ayant pourtant un niveau d’expression estimé sur Xi égal à 21 % de celui du Xa.
Sur les 299 gènes du X analysés par Talebizadeh et al. (2006), seuls 9 ont montré une surexpression moyenne chez les femmes d’au moins 50 % dans au moins trois des onze tissus étudiés. Parmi eux, cinq avaient échappé à l’inactivation dans 8 ou 9 des lignées hybrides de Carrel et Willard, mais deux n’y avaient échappé dans aucune et un n’y avait échappé que dans 2 lignées sur 9 (le dernier n’avait pas été testé par eux). L’article pointe certaines tendances congruentes apparues pour une douzaine de gènes, mais constate que « globalement, aucun profil cohérent n’a été observé entre les résultats produits par (1) le système hybride de cellules somatiques, (2) l’expression dans les fibroblastes de femmes et (3) notre calcul des ratio F/H du niveau d’expression dans onze tissus ».
Pour les auteur·es, les recherches doivent être poursuivies notamment en employant des techniques plus précises de mesure de l’expression des gènes, mais leur étude conforte pour l’instant l’idée qu’au final, le niveau d’expression des gènes du X (et des autosomes) « est globalement similaire entre femmes et hommes », comme venaient déjà de l’indiquer Nguyen & Disteche (2006) [21]. L’étude vient aussi conforter l’idée que le niveau d’expression des gènes du X est grandement influencé par d’autres facteurs que le seul processus d’inactivation du X.
2008 : confirmation de la portée limitée des résultats de Carrel et Willard
Deux ans plus tard, Johnston et collègues (2008) publient dans PLOS Genetics une étude qui revient sur cette question en utilisant la même méthode que Talebizadeh et al. (2006). En effet, écrivent les auteur·es, la technique utilisée par Carrel et Willard peut détecter un faible niveau d’expression d’un gène sur le Xi et le déclarer échappant à l’inactivation, mais on ne sait pas si au final, son niveau global d’expression diffère significativement entre femmes (Xa + Xi) et hommes (un seul X).
Dans cette étude, les ratios d’expression F/H ont été calculés sur les 371 gènes du X et 11 952 gènes d’autosomes exprimés dans des lignées cellulaires de lymphocytes « immortalisés » provenant de 210 sujets issus de quatre populations : des Etats-uniens originaires du nord-ouest de l’Europe (CEU), des Chinois Han de Pékin (CHB), des Japonais de Tokyo (JPT) et des Yorubas d’Ibadan au Nigeria (YRI).
Dans chacune de ces populations, le ratio F/H moyen d’expression des gènes du X était supérieur à 1, mais de très peu, représentant un surcroît d’expression chez les femmes de 2.6 %, 3.4 %, 1.5 % et 2.2 % respectivement pour CEU, CHB, JPT et YRI. « Les gènes échappant à l’inactivation du X n’ont donc pas d’effet important sur le niveau global d’expression des gènes du X dans les lignées de cellules féminines », et cela indique que la compensation femmes-hommes du dosage en gènes du X « est efficace », commentent les auteur·es.
Les auteur·es ont cherché à identifer les gènes responsables de ce surcroît moyen d’expression chez les femmes des gènes du X, certes léger mais observé de manière régulière – et propre au X, les gènes des autosomes n’ayant en moyenne pas montré de différence significative d’expression entre femmes et hommes. L’article rapporte que sur les quatres populations regroupées, seuls 4 % des gènes du X avaient un surcroît moyen d’expression statistiquement significatif à p<0.05 et 3 % à p<0.001 (sans correction pour comparaisons multiples), soit 15 ou 11 gènes respectivement. En tenant compte de toutes les différences F-H même non statistiquement significatives, seuls 20 gènes [22] du X (soit 5.4 % des 371 testés) avaient un niveau moyen d’expression supérieur dans les quatre populations à la fois, supérieur d’ « environ 50 % » pour les 6 gènes les plus surexprimés mais de « beaucoup moins pour la plupart des autres » [23].
Sous l’hypothèse que ces 20 gènes étaient en moyenne surexprimés chez les femmes parce qu’ils échappaient à l’inactivation, cela suggère selon les auteur·es que « les gènes échappant à l’inactivation du chromosome X ont une contribution potentielle aux différences phénotypiques entre les sexes plus limitée que ce qu’on a cru un temps ». En outre, la variabilité du niveau d’expression de ces gènes ne différait pas significativement entre femmes et hommes : « nous en concluons que les gènes qui échappent à l’inactivation du X dans ces lignées cellulaires ne sont pas exprimés de manière plus variable chez les femmes que chez les hommes ».
Enfin, l’article rapporte également que 12 des gènes de PAR1 inclus dans l’étude ne présentaient pas de différence statistiquement significative d’expression entre femmes et hommes (les données en ligne montrent qu’il en était de même pour le gène SYBL1 de PAR2), la seule exception trouvée étant pour le gène CD99 de PAR1, significativement moins exprimé chez les femmes quoique faiblement (de – 3.5% à – 6.1 % selon la population).
Les auteur·es soulignent que les résultats des études qui ont porté sur l’échappement au processus d’inactivation du X varient considérablement en termes de nombre et d’identité des gènes concernés, ce qui pourrait être en partie expliqué par la variabilité de ce phénomène selon les tissus. Cependant, « il est également possible que la variabilité inter-individuelle d’expression non liée à ce phénomène augmente le risque de détections de faux positifs ou faux négatifs dans les petits échantillons ».
D’autres études telles que Zhang et al. (2013) [24] et Cotton et al. (2013)[25] viennent ensuite confirmer qu’hors régions PAR, beaucoup moins que 15 % des gènes du X échappent systématiquement à l’inactivation – peut-être 0 tout simplement, hormis le gène Xist dont l’activation systématique sur Xi a justement pour effet d’activer le mécanisme d’inactivation. Dans toutes les études faites sur ce sujet, le classement d’un gène dans la catégorie « échappe », « échappe variablement » ou « soumis » à l’inactivation dépend en fait de seuils parfaitement arbitraires, le niveau moyen d’expression des gènes portés par le Xi suivant en réalité un continuum et présentant une grande variabilité inter-individuelle, comme le montre par exemple le graphique reproduit ci-dessous.
En outre, les données disponibles suggèrent que l’échappement partiel et variable des gènes du Xi à l’inactivation se traduit au final par une différence entre femmes et hommes assez modeste en termes de niveau global d’expression des gènes du chromosome X, très faiblement sur-exprimés chez les premières hormis pour quelques-uns qui atteindraient un niveau d’expression environ 50 % supérieur. Par ailleurs, on ne sait pas dans quelle mesure les effets biologiques de cette différence sont éventuellement neutralisés ou compensés par les autres différences entre femmes et hommes issues de l’expression des gènes du Y. Enfin, on ne sait pas non plus dans quelle mesure cette différence est influencée par les vécus différents des femmes et des hommes.
Le chiffre clé issu de Carrel & Willard (2005) va néanmoins continuer à être pris pour acquis, et indûment interprété par Peggy Sastre puis par Claudine Junien.
Peggy Sastre enfonce néanmoins le clou, puis Claudine Junien arrive en renfort
En 2009, dans son livre intitulé Ex Utero. Pour en finir avec le féminisme, Peggy Sastre copie-colle une partie du billet du site anonyme cité plus haut, dont sa traduction de la phrase choc : « Il n’y a pas un génome humain, mais bien deux : le génome mâle et le génome femelle ». Histoire de lui donner encore plus de poids, elle prétend que cette phrase a été écrite par Willard « dans Nature » alors qu’elle est tirée d’un communiqué de presse [26].
C’est trois ans plus tard que la généticienne Claudine Junien fait son entrée dans le débat public francophone sur les différences femmes-hommes, via la publication d’une synthèse en français dans la revue Médecine/Sciences (Junien et al. 2012). Intitulé « Le dimorphisme sexuel au XXIè siècle », l’article porte en exergue une citation en anglais qui donne le ton : « Si ce n’était pour le sexe, une grande partie de ce qui est flamboyant et beau dans la nature n’existerait pas. Les plantes ne fleuriraient pas. Les oiseaux ne chanteraient pas. Les cerfs ne verraient pas leurs bois pousser. Les cœurs ne battraient pas si vite » (ma traduction).
Attribuée au « Dr. Tatiana » et tirée d’une synthèse sur « le dimorphisme sexuel » publiée dans Nature Reviews Genetics en 2009, cette citation vient du livre Dr. Tatiana’s sex advice to all creation (2002) de la journaliste scientifique Olivia Judson. Hasard ou pas, lorsqu’on la cherche en anglais dans Google, la première reprise de cette citation est trouvée dans le livre de théologie morale de Christopher Chenault Roberts, alors récent converti au catholicisme romain : Creation and covenant: the significance of sexual difference in the moral theology of marriage (2007, p. 242).
De manière également peu banale, l’article de Junien et al. (2012) se termine par un remerciement d’autant plus remarquable par sa tournure personnelle que Claudine Junien (« mariée, deux enfants », précise-t-elle dans son CV) n’en est pas la seule auteure : « Claudine Junien remercie Jean-Claude Kaplan pour ses précieux conseils, ses encouragements, et son indéfectible soutien à la cause du sexe biologique. » Cet article vise bien en effet à défendre « la cause du sexe biologique », au sens de l’idée que le sexe biologique correspond à une variable biologique bien identifiée, et qu’elle distingue fondamentalement les hommes des femmes comme les mâles des femelles des autres espèces sexuées (ou l’inverse), et ce y compris sur le plan cognitif et comportemental.
Piochant indistinctement dans les données de la biologie humaine et celles concernant d’autres espèces (principalement souris et rats de laboratoire), en s’abstenant au besoin de le signaler, l’article est significativement encore introduit ainsi : « Dès l’instant de notre conception, chacun d’entre nous est doté d’un sexe biologique. Sur les 23 paires de chromosomes héritées de nos parents, c’est la paire de chromosomes sexuels qui fait toute la différence, XX pour une fille et XY pour un garçon. Il n’existe que de très rares cas (0,018 % des naissances) pour lesquels le sexe, à la naissance, peut être ambigu ». Deux références sont citées à l’appui de ce court passage : McCarthy et Arnold (2011) sur la « différenciation sexuelle du cerveau » et Sax (2002) sur la fréquence de l’intersexuation, un classique de la littérature « anti-gender ».
Lu au pied de la lettre, ce passage est presque correct : après tout, on peut dire que nous naissons tous·tes dotés d’un sexe biologique, que celui-ci soit femelle, mâle ou indéterminé. Mais on se demande pourquoi les auteures ont choisi d’invisibiliser ici les quelques 0.25 % d’humains qui ne sont ni XX, ni XY (Genetics Home Reference 2017), ainsi que toutes les autres personnes intersexes qui ne présentent pas à la naissance une ambiguité des organes génitaux externes (et non du sexe) – ou plutôt on ne se le demande pas, et nous verrons plus loin pourquoi il était important de souligner ce détail.
L’article a tout ce qu’il faut pour plaire à Peggy Sastre, qui en publie avec l’aimable autorisation des auteures une version légèrement vulgarisée dans Science… & pseudo-sciences (Sastre 07/2014), revue de scepticisme scientifique instrumentalisée à l’occasion par Nicolas Gauvrit, Franck Ramus et elle-même au service de la « cause du sexe biologique ». On remarque qu’elle rend encore plus trompeur le passage ci-dessus, conçu pour donner l’illusion que la binarité du sexe biologique est presque parfaite chez l’humain : « […] XX pour une fille et XY pour un garcon. Il n’existe que de très rares cas (0,018 % des naissances) pour lesquels le sexe peut être ambigu » (elle a supprimé « à la naissance »).
Deux mois plus tard, Peggy Sastre passe au grand braquet et réalise un beau coup, avec sa publication d’un livre préfacé par Martin Winckler, médecin préféré des féministes françaises : Le sexe des maladies, plaidoyer pour une « médecine différenciée selon le sexe » que j’ai déjà brièvement évoqué [27] (Sastre 2014). En tant que chroniqueuse pour Slate.fr, elle a droit à une publicité sur le site sous la forme d’une citation d’un extrait (Sastre 11/09/2014). Cela commence ainsi : « Certaines différences sexuelles sont élémentaires et manifestes : les femmes ont un vagin, les hommes un pénis » (peut-être faudrait-il prévenir Peggy qu’elle a un clitoris ?). Suit très vite la description de la différence « nichée au cœur de nos cellules » qui est « la plus fondamentale » : « les femmes ont deux chromosomes X quand les hommes ont un X et un Y […]. Dès lors, les gènes des hommes et des femmes ne sont pas tous identiques, une réalité qu’Huntington Willard, l’un des 250 coauteurs de l’annotation du chromosome X [28], résume en ces termes : « Il n’y a pas un génome humain, mais bien deux : le génome mâle et le génome femelle ». Bis repetita placent.
Après la promotion des écrits de Claudine Junien par Peggy Sastre, c’est maintenant à la première de promouvoir ceux de cette dernière. Fin 2014, sur la page du programme « Cœur des femmes » d’une fondation dont Claudine Junien est membre du conseil scientifique, il est signalé qu’elle soutient la « jeune journaliste » Peggy Sastre, qu’elle a été interviewée dans Télématin sur France 2, et qu’elle y est revenue « sur l’importance d’une médecine différenciée et sur les gènes qui constituent l’homme et la femme. Ainsi, il n’y a pas un génome humain, mais bien deux : le génome mâle et le génome femelle. » (Fondation Recherche Cardio-Vasculaire 22/12/2014, en gras sur le site).
Alors que Claudine Junien n’avait auparavant jamais mobilisé ces éléments de langage, ni même l’estimation par Carrel & Willard (2005) de 15 % à 25 % des gènes du X échappant à l’inactivation – que ce soit dans Junien et al. (2012), dans une autre synthèse sur « le dimorphisme sexuel » publiée par les mêmes auteures (Gabory et al. 2009), ou encore dans les paragraphes consacrés au même sujet dans Junien (2011) [29] et Scheen & Junien (2012) [30] –, elle va désormais s’en servir sans modération.
Le fameux argument arrive dans la bouche de Claudine Junien
C’est début décembre 2015 qu’est véritablement donné le coup d’envoi de la série d’interventions publiques de Claudine Junien destinées à promouvoir une « médecine différenciée selon le sexe » – ainsi que les écrits de Peggy Sastre, tantôt en solo, tantôt en duo avec cette dernière. Elle donne le 1er décembre une conférence à l’Académie des sciences (Junien 12/2015), dans le cadre d’une journée sur le thème du « dimorphisme sexuel » commune avec l’Académie de médecine. Elle en est la co-organisatrice, en tant qu’animatrice d’un groupe de réflexion sur le thème des différences entre les sexes qui regroupe les deux Académies. Voici ce que la professeure émérite de génétique médicale et membre de l’Académie de médecine explique sur « les origines génétiques du dimorphisme sexuel », commentant une diapositive qui montre « 0.1 % » entre Barack Obama et Vladimir Poutine, « 1.5 % » entre Barack Obama et Angela Merkel, et « 1.5 % » entre Vladimir Poutine et un chimpanzé affublé d’une chemise et d’une cravate (2’50’’) :
« Cette diapositive vous montre le poids de la génétique. Quelle est la différence entre deux hommes de la même espèce ? Eh bien entre ces deux personnages, comme entre tous les hommes, il y a à peu près 0.1% de différence génétique. Mais si on regarde les différences entre un homme et une femme, eh bien il y a 15 fois plus de différence, donc c’est une différence qui est énorme, et ce qui est intéressant, c’est qu’elle est du même ordre de grandeur qu’entre deux mâles d’espèces différentes, ici le chimpanzé et l’homme. Et ces différences génétiques vont aboutir à des différences encore plus importantes en termes d’expression de gènes gérée par l’épigénétique, puisqu’on a à peu près 40 % des gènes, en moyenne, qui sont différentiellement exprimés, et c’est ce qu’on retrouve aussi entre le chimpanzé et l’homme, et en particulier dans le cerveau. »
Elle donne ensuite les éléments du calcul de la différence entre femmes XX et hommes XY : « les chromosomes X contiennent environ 1400, 1500 gènes [1400 sur la diapositive], et il y a 15 à 25 % de ces gènes qui […] vont échapper à l’inactivation de l’X, donc il y a certains gènes qui vont être plus exprimés chez une femme que chez un homme ». Quant au chromosome Y, il lui reste « à peu près une centaine de gènes [“< 100” sur la diapositive], mais il y a en particulier 17 gènes régulateurs qui sont très conservés à travers l’évolution, et qui sont spécifiques du mâle, mais surtout on retrouve sur le chromosome X des paralogues qui sont précisément des gènes qui échappent à l’inactivation de l’X. Or les paralogues sur l’X et le paralogue correspondant sur l’Y forment des paires qui sont semblables, qui sont très proches, mais qui ne sont pas strictement identiques ».
Sur une diapositive montrée ensuite (7’11’’), la différence génétique qui dure « tout au long de la vie » est résumée en deux points : la différence de « séquence » égale à « 1.5 % » issue des hétérochromosomes, et une différence d’expression des gènes des autosomes égale à « 40 % ». Pour ce qui est de l’impact sur les pathologies humaines, Claudine Junien renvoie au livre de Peggy Sastre qui selon elle « montre que puisqu’on a des différences, eh bien, les maladies ont un sexe », sa diapositive se résumant à la couverture dudit livre.
Pour bien comprendre le contexte de ces propos au-delà de la promotion d’une médecine différenciée selon le sexe, signalons qu’en guise de conclusion, Claudine Junien appelle plus généralement à tenter de démêler les rôles respectifs du sexe et du genre, ce qui est louable, mais en fixant à ces recherches des objectifs bien particuliers (21’30’’) : « Et grâce à ça, on devrait pouvoir avoir une meilleure utilisation des dispositions naturelles des uns et des autres, ou des unes et des autres, et trouver des nouvelles bases pour la parité. Et puis dans la société, dans la vie privée, ça permettrait peut-être aussi d’optimiser les relations et la communication entre les hommes et les femmes, qui se connaîtraient mieux grâce à ces pratiques » (fin de l’exposé). Lors des questions, elle enfoncera bien le clou en exprimant sa conviction que quoi qu’on fasse, un cerveau reste féminin si on est née XX et masculin si on est né XY, d’où un risque d’ « incompatibilité » entre sexe et genre chez les personnes qui changent de sexe [31]. Comme l’indique le titre du document rédigé en lien avec cette conférence, il s’agit bien d’un « plaidoyer », et ce notamment contre un certain féminisme [32].
Déploiement de l’argument choc
Suite à cette journée de décembre et aux conclusions du groupe (temporaire) de réflexion, l’Académie de médecine émet en juin 2016 une note d’information plaidant pour la prise en compte des « différences biologiques entre les sexes » dans la recherche scientifique et la médecine (Acad. Nat. Méd. 06/2016). Tenant sur deux courtes pages, la note fait la part belle à l’argument choc dans un paragraphe qualifiant d’ « obsolète » la « vision actuelle sur la différenciation du sexe », points de suspension suggestifs à l’appui : « En effet, si la ressemblance du génome, en termes de séquence, entre 2 hommes ou 2 femmes est de 99,9%, la ressemblance entre un homme et une femme n’est que de 98,5%, du même ordre de grandeur qu’entre un humain et un chimpanzé, de même sexe… ».
L’affirmation est reprise sans sourciller sur le site PourquoiDocteur (Guerin 24/06/2016) [33], sur un blog de L’Express tenu par un journaliste spécialisé en santé (Olivier 26/06/2016) [34], ou encore dans la Revue Médicale Suisse (Nau 07/2016) [35]. On la retrouve en septembre dans les pages santé du Figaro (Court & Thibert 02/09/2016) [36], ce qui alimente la sphère anti-gender française (Dickès [16/09/2016] [37]) et hispanophone (Actuall repris ensuite sur le site chilien Temas & Noticias [Lozano 08/09/2016]) [38].
Claudine Junien réinjecte l’argument choc lorsqu’elle relance la machine médiatique en février 2017 : elle l’emploie dans un opus édité par l’Institut Diderot (Junien 04/02/2017) [39], sur PourquoiDocteur dans le cadre de l’émission « La médecine au féminin » du Dr Sophie Lemonier (Lemonier 18/02/2017) [40], ou encore dans une tribune pour Le Figaro (Junien 10/02/2017) [41]. Elle l’utilise également dans un article censé présenter des « faits biologiques irréfutables » pour défendre « une recherche et une médecine sexuellement différenciées », mais aussi et surtout pour faire admettre les différences que « sous prétexte de parité, on a trop longtemps évité en France de reconnaître […], au nom de l’égalité mais au mépris des évidences scientifiques » : « le sexe biologique a trop longtemps été nié dans notre pays au profit de la primauté du genre, au nom de l’égalité des sexes » (Junien & Gauvrit 26/02/2017) [42]. Signé dans The Conversation avec Nicolas Gauvrit et également publié en tant que communiqué de presse sur le site de l’INRA, ce texte semble être surtout de la plume de Peggy Sastre et Claudine Junien. En effet, un texte parfaitement identique est publié le lendemain sur Atlantico.fr avec leurs seuls deux noms de contributrices (et il est précisé à la fin de celui de The Conversation : « Peggy Sastre, auteure de “Le sexe des maladies” (Éditions Favre 2014), et de “La domination masculine n’existe pas” (Éditions Anne Carrière 2015), nous a apporté une large contribution pour la réalisation de cet article et nous l’en remercions chaleureusement. »).
Une sorte d’acmé est atteinte avec le texte que Junien et Sastre (04/04/2017) co-signent dans Causeur, dont le titre (« Santé : on naît femme, on ne le devient pas ») affiche clairement la volonté des auteures d’attaquer de front le féminisme universaliste représenté par Simone de Beauvoir, comme Claudine Junien l’avait déjà fait deux mois plus tôt [43]. A force d’être rabâchées, leurs idioties se téléscopent et fusionnent en une : l’écart entre « un homme et une femme » est désormais « 15 fois plus important qu’entre un humain et un chimpanzé de même sexe ! » [44] Ca décoiffe, et la mouvance anti-gender adore car c’est imparable, puisque venant de « deux femmes, professeur de Génétique médicale et docteur en philosophie des sciences » (La Manif Pour Tous 19/04/2017 ; Enseignants pour l’enfance 24/04/2017 [45]).
Il est grand temps que ces billevesées cessent de circuler en passant pour être des faits scientifiques.
PARTIE 3 – POURQUOI C’EST MULTIPLEMENT FAUX ET INEPTE
Calcul de la « différence génétique » entre femmes et hommes
Faisons dans un premier temps comme si le calcul de la différence génétique entre femmes et hommes avait un sens, comme si l’expression (quel que soit son niveau) d’une seconde copie d’un gène était équivalente à la possession d’un gène différent supplémentaire, et comme s’il y avait effectivement 15 % des gènes du X qui échappent à l’inactivation.
Rappelons qu’à l’appui de l’estimation de 1.5 % de différence entre femmes et hommes, Claudine Junien répète en 2015, 2016 et 2017 qu’il y a environ 23 000 gènes au total dans le génome humain, 100 gènes sur le chromosome Y, 1500 sur le X (sans indiquer les références de ces estimations), et que 15 % de ces derniers, soit 225 gènes, échappent à l’inactivation.
Puisqu’avec 225 gènes on ne trouve pas 1.5 % de différence, je suppose qu’elle a plutôt utilisé l’estimation de 250 gènes faite par Willard en 2005 (soit « 200 à 300 gènes » comme repris dans Sastre [2009]). Pour obtenir 250, Willard avait retenu l’estimation de 1000 gènes dont 25 % échappant à l’inactivation, et non de 1500 dont 15 % échappant, mais Claudine Junien se fiche sans doute de ce genre de détails : l’essentiel était de se débrouiller pour retomber sur environ 1.5 % de différence.
Elle se fiche de même de s’assurer que l’estimation de 15 % de gènes du X échappant à l’inactivation est correcte et ne prend manifestement pas non plus la peine de lire les références qu’elle cite. En effet, la seule fois où elle cite une référence précise à l’appui de cette estimation, elle affirme qu’ « il s’est avéré que 10 à 15 % des gènes échappaient à cette inactivation chez la femme » en citant en référence « Carolyn J. Brown 1991 » (Junien 04/02/2017). Or cela était loin d’être « avéré » en 1991 comme on l’a vu, et par ailleurs Carolyn J. Brown n’a publié aucun article seule en 1991. Cette référence mal recopiée est sans doute celle de l’article de Brown et collègues (dont Willard) dans lequel a été rapportée la découverte d’un nouveau gène, baptisé Xist par les auteur·es (Brown et al. 1991). Il rapportait le constat que ce gène ne s’exprimait que sur le chromosome X inactivé, et suggérait (à raison) qu’il était au cœur du processus d’inactivation du X.
Le calcul à la louche de Claudine Junien est donc le suivant :
- 100 / 23 000 = 0.43 % de différence crée par les gènes spécifiques au Y,
- 250 / 23 000 = 1.09 % de différence crée par les gènes « spécifiques » au X inactivé,
soit 1.5 % de « différence génétique » au total.
Tout d’abord, nous supposerons que lorsque Claudine Junien dit « gènes », elle veut dire « gènes codants », conformément à la définition passablement datée qu’elle a encore donnée récemment : « La génétique s’intéresse aux gènes – 23 000 gènes, des segments d’ADN associés à des protéines […] répartis sur 46 chromosomes » (Junien 04/02/2017). En effet, dans le cas contraire le nombre total de gènes ne serait pas de l’ordre de 23 000, mais plutôt de 43 000 (pour cette estimation et les suivantes, voir la Figure 1), ce qui ramènerait déjà la différence à moins de 0.8 %.
Une estimation actuelle plus plausible du nombre de gènes codants est de 841 pour le X, 63 pour le Y et 20 300 gènes dans le génome haploïde humain. Par ailleurs, comme les 19 gènes des régions PAR sont les mêmes sur le X et le Y, il n’est pas logique de les compter dans le calcul des différences. Cela ramène le nombre de gènes spécifiques au Y à 44 (au lieu de 100), et le nombre de gènes « spécifiques » au X inactivé à 15 % de 841 moins 19, soit 107 gènes (au lieu de 250). En utilisant ces estimations, la différence devient égale à (44 + 107) / 20 300, soit 0.74 % de différence entre personnes XX et XY, et non 1.5 %.
En outre, il n’est pas logique de compter les deux chromosomes de la paire XY au numérateur sans compter aussi au dénominateur les seconds chromosomes des paires d’autosomes, puisque comme le second X, ils portent eux aussi une seconde copie de gènes, et elle n’est pas soumise à un mécanisme d’inactivation. C’est donc le nombre total de gènes (codants) présents dans le génome diploïde qu’il faudrait compter au dénominateur, soit environ 39 700 pour simplifier [46]. Avec cette nouvelle correction, la différence devient égale à (44 + 107) / 39 700, soit 0.38 %.
Prise en compte des niveaux d’expression
Cela étant, est-il correct de compter pour 1 chacun des gènes du X échappant à l’inactivation alors que leur niveau d’expression sur le X inactivé est souvent faible ? De fait, bien que Claudine Junien parle de différence en termes de « séquence d’ADN », la logique de son calcul n’est pas strictement génomique puisque la différence calculée repose déjà pour plus des deux tiers sur la prise en compte d’un niveau d’expression. En effet, comme on l’a vu les gènes comptés comme « échappant à l’inactivation » sont ceux ayant un niveau d’expression supérieur à un seuil fixé arbitrairement sur le X inactivé, et ce dans un pourcentage des échantillons testés supérieur à un seuil également fixé arbitrairement. Puisqu’elle tient compte du niveau d’expression des gènes du X, il faut le faire vraiment et appliquer la même logique à tous.
Idéalement, il faudrait donc compter non pas le nombre de gènes du X « échappant à l’inactivation », mais le nombre total de gènes du X pondéré par la différence de niveau d’expression de chacun observée en moyenne entre personnes XX et personnes XY. Si on se fie à Talebizadeh et al. (2006) et à Johnston et al. (2008) au moins, cette différence moyenne semble ne pas pouvoir être supérieure à 5 %. Avec cette estimation, ce serait tout au plus 42 « équivalents gènes » (5 % de 841) qu’il faudrait compter pour calculer la « différence génétique » due aux gènes « spécifiques » au X inactivé [47]. Avec cette nouvelle correction, la différence deviendrait égale à environ (44 + 42) / 39 640, soit 0.22 % de différence entre personnes XX et XY, et non 1.5 %.
On pourrait objecter qu’il faudrait aussi tenir compte des niveaux d’expression des gènes du X hors regions PAR relativement aux gènes des autosomes, supérieur au moins pour certains des gènes échappant à l’inactivation (Pessia et al. 2012 ; Pessia et al. 2014), ce qui ferait remonter la différence F-H. On pourrait objecter a contrario que puisqu’on compte des gènes du X ayant une seconde copie qui s’exprime, il faudrait aussi ajouter au dénominateur les copies des gènes des autosomes dont plusieurs copies sont communément présentes et s’expriment [48], ce qui ferait diminuer la différence. Mais si on va par là, il faut compter aussi les copies multiples de certains gènes du Y, ce qui va la faire remonter. Arrêtons-nous là : ces considérations sont vaines, car ce qui compte au final, ce sont les effets concrets de ces différences génétiques.
Il est clair que vis-à-vis des objectifs de Claudine Junien de « trouver des nouvelles bases pour la parité », « d’optimiser les relations et la communication entre les hommes et les femmes » et de développer une « médecine sexuellement différenciée » pour les maladies communes aux femmes et aux hommes, certaines de ces différences génétiques n’ont à coup sûr aucune pertinence. Ainsi, comme on l’a vu plus haut, sur les 44 gènes du Y restant dans le calcul ci-dessus, une quinzaine seulement s’expriment ailleurs que dans les testicules ou les spermatozoïdes. A contrario, la différence d’un seul gène du Y, à savoir Sry, cause à coup sûr des différences anatomiques et physiologiques non négligeables. L’affirmation qu’il y a 1.5 % de différence génétique entre femmes et hommes est accompagnée par Claudine Junien, Peggy Sastre et à l’occasion Nicolas Gauvrit d’autres propos plus que discutables sur les conséquences de ces différences, ce qui mérite de faire l’objet de critiques ad hoc. Revenons pour l’instant à la « différence génétique », et finissons-en en examinant la pertinence des comparaisons faites avec la différence entre deux humains de même sexe et avec la différence entre humain et chimpanzé.
Comparaison avec la différence entre deux humains de même sexe
Comme on l’a vu plus haut, la différence génétique moyenne entre deux êtres humains de même sexe n’est pas connue avec précision, mais est actuellement estimée à environ 0.5 % si on tient compte non seulement des environ 0.1 % en termes de SNP (ces différences étant de même nature que celles constituées par les gènes spécifiques au Y), mais aussi des environ 0.4 % en termes de CNV (ces différences étant de même nature que celles constituées par les gènes du X échappant à l’inactivation). Même en prenant au sérieux l’estimation fausse et absurde de 1.5 % de différence entre femmes est homme, celle-ci serait donc trois fois plus grande qu’entre deux humains de même sexe, et non quinze fois plus.
Par ailleurs, outre que le calcul de la différence femmes-hommes est spécieux, la comparaison de ces deux pourcentages l’est aussi car ils ne sont pas du tout calculés de la même manière. En effet, les 0.5 % de différence entre deux humains de même sexe sont calculés en nombre de bases différentes, et non en nombre de gènes. Si on appliquait la même logique (absurde) aux différences entre humains de même sexe, on obtiendrait une différence énorme (20, 30, 40 %, peut-être même plus), car les SNP sont répartis dans tout le génome.
Enfin, si Claudine Junien appliquait de manière cohérente sa conception du sexe « génomique » à tous les êtres humains, elle devrait de même soutenir qu’il existe environ 1 % de différence entre une femme XX et une femme X0, et de même entre un homme XY et un homme XXY, ou encore sans doute entre une femme XXX et une femme XX. Elle devrait par conséquent dénoncer aussi, par exemple, le scandale constitué par le fait de considérer qu’une personne XXY est un homme comme les autres, ou encore par le fait de considérer qu’un homme trisomique 21 est un homme comme les autres, puisqu’il y a environ 235 gènes codants sur le chromosome 21, soit à nouveau environ 1 % de différence génétique si on calcule comme elle. Mais Claudine Junien ne soutient rien de tout cela et ne plaide par pour une médecine « différenciée selon le caryotype » : ce qui la motive, c’est « la cause du sexe biologique » au sens d’un sexe parfaitement binaire, et pour rester focalisée sur cette cause elle préfère (comme on l’a vu) faire comme si les variations de caryotype n’existaient tout simplement pas.
Comparaison avec la différence entre humain et chimpanzé
Pour ce qui est de la différence entre un humain et un chimpanzé « de même sexe », on a vu qu’elle était non pas égale à 1.5 % comme le martèle Claudine Junien en reprenant un chiffre périmé depuis 2001, mais au bas mot de 4 à 5 %. Même en prenant au sérieux l’estimation fausse et absurde de 1.5 % de différence entre femmes est homme, celle-ci serait donc au moins trois fois plus petite qu’entre humain et chimpanzé, et non égale.
En outre, à l’instar de la différence entre deux humains de même sexe, elle est calculée en nombre de bases et ne peut donc être comparée avec une différence femmes-hommes calculée en nombre de gènes.
Par ailleurs, comme on l’a vu, cette estimation de 4 à 5 % ne tient pas compte des vastes réarrangements génomiques dont on ignore les conséquences, et en tout état de cause les différences anatomiques, physiologiques ou encore cognitives entre humain et chimpanzé sont manifestement incommensurables aux différences existant en moyenne entre deux humains, quel que soit leur sexe.
Enfin ici encore, si Claudine Junien appliquait de manière cohérente sa conception du sexe « génomique » à toutes ces considérations, elle devrait s’abstenir de considérer qu’un chimpanzé de sexe femelle est « de même sexe » qu’une femme et qu’un chimpanzé de sexe mâle est « de même sexe » qu’un homme. En effet, les gènes portés par les chromosomes X et Y des chimpanzés diffèrent substantiellement de ceux portés par les chromosomes X et Y des humains. Cette différence est encore plus grande entre souris et humains, sans compter que Claudine Junien indique elle-même que seuls 3% des gènes du X échappent à l’inactivation chez la souris [49] : elle devrait logiquement en conclure que sur le plan génétique, le « sexe biologique » de la souris est assez différent de celui de l’être humain, or elle plaide au contraire pour l’étude systématique des différences biologiques liées au sexe chez les souris pour révéler celles existant chez l’humain. Mais rappelons encore une fois que ce qui la motive, c’est « la cause du sexe biologique », au sens d’un sexe qui serait non seulement parfaitement binaire dans notre espèce, mais aussi trans-espèces, et pour cela il vaut mieux faire comme si ces différences n’existaient pas.
En conclusion, les propos de Claudine Junien sont basés sur des chiffres faux, des informations périmées et des estimations de seconde main dont elle ne s’est souciée d’examiner ni la solidité, ni la signification concrète, le principe même du calcul de « la différence génétique » entre femmes et hommes est absurde, et les comparaisons faites avec les deux autres pourcentages sont également ineptes à plusieurs titres. Nous avons ici clairement affaire à de la pseudo-science enrôlée au service d’une idéologie, et il est vraiment regrettable qu’elle ait pu atteindre un tel degré de visibilité et de légitimation scientifique.
Odile Fillod
____________________________
Références et éléments de vulgarisation cités
– Acad. Nat. Méd. (06/2016) « Parité en santé : la recherche scientifique et la médecine ne peuvent plus ignorer les différences biologiques entre les sexes », Information, en ligne sur www.academie-medecine.fr/publication100100563/
– BADOC Michel, BAYLE-TOURTOULOU Anne-Sophie (2016) Comment les neurosciences éclairent les décisions d’achat du consommateur, Eyrolles
– BALATON Bradley P. , COTTON Allison M., BROWN Carolyn J. (2015) Derivation of consensus inactivation status for X-linked genes from genome-wide, Biology of Sex Differences, vol.35, https://doi.org/10.1186/s13293-015-0053-7
– BELLOTT Daniel et al. (2014) Mammalian Y chromosomes retain widely expressed dosage-sensitive regulators, Nature, vol. 508, p. 494-499
– BLACKLESS Melanie et al. (2000) How Sexually Dimorphic Are We? Review and Synthesis, American Journal of Human Biology, vol. 12, p. 151-166
– BRITTEN Roy J. (2002) Divergence between samples of chimpanzee and human DNA sequences is 5%, counting indels, PNAS, vol.99, p. 13633-13635
– BROWN Carolyn J., […], WILLARD Huntington (1991) A gene from the region of the human X inactivation centre is expressed exclusively from the inactive X chromosome, Nature, 349, vol. 349, p. 38-44
– BROWN Carolyn J., CARREL Laura, WILLARD Huntington F. (1997) Expression of genes from the human active and inactive X chromosomes, American Journal of Human Genetics, vol.60(6), p. 1333-1343
– CARREL Laura, […], WILLARD Huntington F. (1999) A first-generation X-inactivation profile of the human X chromosome, PNAS, vol. 96(25), p. 14440-14444
– CARREL Laura, WILLARD Huntington F. (2005) X-inactivation profile reveals extensive variability in X-linked gene expression in females, Nature, vol.434(7031), p.400-404
– CHEN Feng-Chi, LI Wen-Hsiung (2001) Genomic divergences between humans and other hominoids and the effective population size of the common ancestor of humans and chimpanzees, American Journal of Human Genetics, vol.68, p. 444-456
– CHENG Ze et al. (2005) A genome-wide comparison of recent chimpanzee and human segmental duplications, Nature, vol.437, p. 88-93.
– CONNOR Steve (17/03/2005) « War of the sexes: How chromosome study shows male and female genetic differences », The Independant
– CONRAD Donald et al. (2010) Origins and functional impact of copy number variation in the human genome, Nature, vol. 464(7289), p. 704-712
– COTTON Allison M., […] BROWN Carolyn J. (2013) Analysis of expressed SNPs identifies variable extents of expression from the human inactive X chromosome, Genome Biology, vol.14(R122), https://doi.org/10.1186/gb-2013-14-11-r122
– COURT Marielle, THIBERT Cécile (02/09/2016) « Médecine : les différences hommes-femmes négligées », Le Figaro
– DAVIES Williams, WILKINSON Lawrence S. (2006) It is not all hormones: alternative explanations for sexual differentiation of the brain, Brain Research, vol. 1126, p. 36-45
– DICKES Jean-Pierre (16/09/2016) « Les hommes et les femmes devraient être soignés différemment. Qu’en pense LGBT ? », www.medias-presse[.]info
– DO O’GOMES Isabelle (17/03/2005) « Les chromosomes X : des atouts considérables pour les femmes », Sciences et Avenir, en ligne sur www.sciencesetavenir.fr/sante
– DOWD Maureen (20/03/2005) « X-celling over men », New York Times, p. 13
– Duke University Medical Center – Duke Institute for Genome Sciences & Policy / MORGAN Kendall (16/03/2005) « Variation in women’s X chromosomes may explain difference among individuals, between sexes », en ligne sur http://genome.duke.edu/press/news/03-16-2005/ et https://www.eurekalert.org/pub_releases/2005-03/dumc-viw031005.php
– ENARD Wolfgang (2016) The molecular basis of human brain evolution, Current Biology, vol.26(20), p. R1109-R1117
– Enseignants pour l’enfance (24/04/2017) « Le “genre” en biologie : ce serait finalement une erreur grossière ? », www.enseignants-pour-enfance.org/spip.php?article930
– Ensembl v90 (août 2017) : données du Genome Reference Consortium Human Build 38 patch release 11 (GRCh38.p11) et de CHIMP2.1.4 consultées en novembre 2017 sur www.ensembl.org/Homo_sapiens et www.ensembl.org/Pan_troglodytes
– FAUSTO-STERLING Anne (2003) How Sexually Dimorphic Are We? Review and Synthesis – Response, American Journal of Human Biology, vol. 15, p. 115-116
– Fondation Recherche Cardio-Vasculaire (22/12/2014) « La femme n’est pas un homme comme les autres… », http://www.fondation-recherche-cardio-vasculaire.org/la-femme-nest-pas-un-homme-comme-les-autres/
– FRANCHINI Lucia, POLLARD Katherine (2017) Human evolution: the non-coding revolution, BMC Biology, vol.15, art.89, DOI 10.1186/s12915-017-0428-9
– FRYNS J. P. et al. (1995) XYY syndrome and other Y chromosome polysomies. Mental status and psychosocial functioning, Genetic Counseling, vol.6, p.197-206
– GABORY Anne, ATTIG Linda et JUNIEN Claudine (2009) Sexual dimorphism in environmental epigenetic programming, Molecular and Cellular Endocrinology, vol.304, p. 8-18
– GELLMAN Charles, HIGY-LANG Chantal (2003) L’Art du contact. Mieux vivre avec les autres, Editions d’Organisation (préface par Philippe Brenot)
– Genetics Home Reference (2017) : https://ghr.nlm.nih.gov/condition accédé le 11/11/2017
– GILGENKRANTZ Simone (2005) Insubordination et sollicitude du chromosome X humain, Médecine/Sciences, vol. 21(6-7), p. 573-577
– GLAZKO Galina et al. (2005) Eighty percent of proteins are different between humans and chimpanzees, Gene, vol.346, p. 215-219
– GOODFELLOW Peter, […], SHAPIRO Larry J. (1984) The cell surface antigen locus, MIC2X, escapes X-inactivation, American Journal of Human Genetics, vol.36, p. 777-782
– GUERIN Marion (24/06/2016) « Hommes- femmes : la médecine sous-estime les différences », www.pourquoidocteur.fr
– GUNTER Chris (2005) Genome biology: she moves in mysterious ways, Nature, 434, p. 279-280
– GUTERL Fred (28/03/2005) « The truth about gender », Newsweek, vol.145(13), p. 42
– HEIDECKER Bettina et al (2010) The gene expression profile of patients with new-onset heart failure reveals important gender-specific differences, European Heart Journal, vol.31(10), p. 1188-1196
– HGNC (11/2017) : base de référence du Hugo Gene Nomenclature Committee en ligne sur www.genenames.org, dont www.genenames.org/cgi-bin/genefamilies/set/714 pour les régions PAR (accédé le 24 novembre 2017)
– HOTZ Robert Lee (17/03/2005) « Women are very much not alike, gene study finds », Los Angeles Times
– HULL Carrie L. (2003) How Sexually Dimorphic Are We? Review and Synthesis – Letter to the Editor, American Journal of Human Biology, vol. 15, p. 112-115
– JANGRAVI Zohreh et al. (2013), A fresh look at the male-specific region of the Human Y chromosome, Journal of Proteome Research, vol.12, p. 6-22
– JOHNSTON Colette, […] et ROSS Mark T. (2008) Large-scale population study of human cell lines indicates that dosage compensation is virtually complete, PLOS Genetics, http://journals.plos.org/plosgenetics/article?id=10.1371/journal.pgen.0040009
– JORDAN-YOUNG Rebecca (2016[2010]) Hormones, sexe et cerveau, Belin, trad. Odile Fillod
– JUNIEN CLaudine (2011) « Les déterminants précoces de la santé et des maladies : épigénétique et environnement », Bulletin de l’Académie Nationale de Médecine, vol.195(3), p. 511-527 [communication faite en mars 2011 en même temps que la candidature de Claudine Junien à une place de membre titulaire de l’Académie nationale de médecine, 3ème division, section des sciences pharmaceutiques, candidature qui a été acceptée]
– JUNIEN Claudine, GABORY Anne et ATTIG Linda (2012) Le dimorphisme sexuel au XXIe siècle, Médecine/Sciences, vol.28(2), p. 185-192
– JUNIEN Claudine (01/12/2015), « Origines Ancestrales, Développementales, Environnementales du dimorphisme sexuel (DS) : Génétique, épigénétique et hormones » en ligne sur www.youtube.com/watch?v=lgPwKYJfT74
– JUNIEN Claudine (04/02/2017) Le corps, ce grand oublié de la parité, www.institutdiderot.fr
– JUNIEN Claudine (10/02/2017) « Peut-on encore soigner les femmes… comme des hommes ? », Le Figaro.fr
– JUNIEN Claudine, GAUVRIT Nicolas (26/02/2017) « Pour une recherche et une médecine sexuellement différenciées : des faits biologiques irréfutables », The Conversation
– JUNIEN Claudine, SASTRE Peggy (27/02/2017) « Pour une recherche et une médecine sexuellement différenciées : des faits biologiques irréfutables », Atlantico.fr
– JUNIEN Claudine, SASTRE Peggy (04/04/2017), « Santé : on naît femme, on ne le devient pas », Causeur
– KIM Min-Sik, […], PANDEY Akhilesh (2014) A draft map of the human proteome, Nature, vol.509, p. 575-581
– KARKI Roshan et al. (2015) Defining “mutation” and “polymorphism” in the era of personal genomics, BMC Medical Genomics, vol.8, doi : 10.1186/s12920-015-0115-z
– La Manif Pour Tous (19/04/2017) « Santé : On naît femme, on ne le devient pas … », www.lmptcollectif-oise.fr/causeur-sante-on-nait-femme-on-ne-le-devient-pas/
– LARROQUE Damien (30/03/2005) « Génome humain : du nouveau sur le chromosome X », Futura-sciences, en ligne sur www.futura-sciences.com/sante/actualites
– LEMONIER Sophie Dr (18/02/2017) « Médecine : les différences hommes-femmes sous-estimées », www.pourquoidocteur.fr
– LOZANO Javier (08/09/2016) « La medicina contra la Ideología de Género: advierte que se puede cambiar de sexo pero no de cerebro », en ligne sur www.actuall.com/familia/la-medicina-la-ideologia-genero-advierte-se-puede-cambiar-sexo-no-cerebro/ et sur www.temas.cl/2016/09/15/la-medicina-contra-la-ideologia-de-genero-advierte-que-se-puede-cambiar-de-sexo-pero-no-de-cerebro/
– LEE P.A. et al. (2016), Global disorders of sex development update since 2006: perceptions, approach and care, Hormone Research in Pediatrics, vol. 85, p. 158-180
– LEVY Samuel et al. (2007) The diploid genome sequence of an individual human, PLoS Biology, vol.5(10), doi : 10.1371/journal.pbio.0050254
– LINSCHEID Caitlin, PETROFF Margaret G (2013) Minor histocompatibility antigens and the maternal immune response to the fetus during pregnancy, American Journal of Reproductive Immunology, vol.69(4), p. 304-314
– LYON Mary F. (2005) No longer ‘all-or-none’, European Journal of Human Genetics, vol.13, p. 796–797 [publié en ligne le 4 mai 2005, en accès libre sur www.nature.com/articles/5201443.pdf?origin=ppub]
– McCARTHY Margaret M., ARNOLD Arthur P (2011) Reframing sexual differentiation of the brain, Nature Neuroscience, vol.14(6), p. 677-683
– MORIN Hervé (01/09/2005) « L’ADN du chimpanzé révèle une part de l’humain », Le Monde
– NAU Jean-Yves (19/03/2005) « Des généticiens lèvent un coin du voile sur le chromosome X », Le Monde
– NAU Jean-Yves (07/2016) « L’homme n’est plus l’égal biologique et médical de la femme », Revue Médicale Suisse, vol.12(525), p. 1298-1299
– NGUYEN Di Kim, DISTECHE Christine M. (2006) Dosage compensation of the active X chromosome in mammals, Nature Genetics, vol.38(1), p. 47-53
– OLIVIER Vincent (26/06/2016) « Santé: la femme n’est pas un homme comme les autres », en ligne sur blogs.lexpress.fr/le-boulot-recto-verso
– PANG Andy et al. (2010) Towards a comprehensive structural variation map of an individual human genome, Genome Biology, vol.11, https://doi.org/10.1186/gb-2010-11-5-r52
– PENN STATE (03/2005), « Men and women: the differences are in the genes », ScienceDaily, www.sciencedaily.com/releases/2005/03/050323124659.htm
– PERRY George H., […], STONE Anne C. (2007) Diet and the evolution of human amylase gene copy number variation, Nature Genetics, vol.39(10), p. 1256-1260
– PESSIA Eugénie et al. (2012) Mammalian X chromosome inactivation evolved as a dosage-compensation mechanism for dosage-sensitive genes on the X chromosome, PNAS, vol.109(14), p.5346-5351
– PESSIA Eugénie, ENGELSTÄDTER Jan, MARAIS Gabriel (2014) The evolution of X chromosome inactivation in mammals: the demise of Ohno’s hypothesis? Cellular and Molecular Life Sciences, vol.71, p.1383-1394
– POWERS Matthew et al. (2017) From sexless to sexy: why it is time for human genetics to consider and report analyses of sex, Biology of Sex Differences, vol.8(15), https://doi.org/10.1186/s13293-017-0136-8
– PREUSS Todd M. (2012) Human brain evolution: from gene discovery to phenotype discovery wrote, PNAS, vol. 109 (suppl.1), p. 10709-10716
– PRÜFER Kay […] et PÄÄBO Svante (2012) The bonobo genome compared with the chimpanzee and human genomes, Nature, 486, p. 527-531
– REDON Richard et al. (2006) Global variation in copy number in the human genome, Nature, vol.444, p. 444-454
– REICH Jens (2001) « Les frontières de l’humain », in Jean-François Mattei (dir.) Le génome humain, Editions du Conseil de l’Europe
– RICHARDSON Sarah S. (2010) Sexes, species, and genomes: why males and females are not like humans and chimpanzees, Biology & Philosophy, vol.25(5), p. 823-841
– RICHARDSON Sarah S. (2012) Sexing the X: how the X became the “female chromosome”, Signs, vol.37(4), p. 909-933.
– ROSS Mark T. et al. (2005), The DNA sequence of the human X chromosome, Nature, vol.434(7031), p.325-337
– ROTIMI Charles N., JORDE Lynn B. (2010) Ancestry and disease in the age of genomic medicine, New England Journal of Medicine, vol.363, p. 1551-1558
– RTFlash (24/03/2005) « Les gènes du chromosome X féminin diffèrent de ceux du X des hommes », en ligne sur www.rtflash.fr [source : « news Nature »]
– SASTRE Peggy (2009) Ex Utero. Pour en finir avec le féminisme, La musardine
– SASTRE Peggy (07/2014) « Les différence sexuelles au XXIè siècle », Science… & pseudo-sciences, n°309
– SASTRE Peggy (2014) Le sexe des maladies, Favre
– SASTRE Peggy (11/09/2014) « Les maladies ont un sexe », Slate.fr
– SAX Leonard (2002) How common is ‘intersex’? A response to Anne Fausto-Sterling, The Journal of Sex Research, vol. 39(3), p. 174-178
– SCHEEN André J., JUNIEN Claudine, (2012) Épigénétique, interface entre environnement et gènes : rôle dans les maladies complexes, Revue Médicale de Liège, vol.67, p. 250-257
– SERAJEE, F. J., HUG, A. H. M. M. (2009) Association of Y chromosome haplotypes with autism, Journal of Child Neurology, vol.24, p. 1258-1261
– SHAPIRO Larry J. et al. (1979) Non-inactivation of an X-chromosome locus in man, Science, vol. 204, p. 1224-1226
– SKALETSKY Helen et al. (2003) The male-specific region of the human Y chromosome is a mosaic of discrete sequence classes, Nature, vol.423(6942), p. 825-837
– SUDMANT Peter et al. (2013) Evolution and diversity of copy number variation in the great ape lineage, Genome Research, vol.23, p.1373-1382
– SUDMANT Peter et al. (2015) An integrated map of structural variation in 2,504 human genomes, Nature, vol.526, p.75-81
– TALEBIZADEH Zohreh, SIMON Stephen D., BUTLER Merlin G. (2006) X chromosome gene expression in human tissues: male and female comparisons, Genomics, vol. 88(6), p. 675-681
– TARTAGLIA N. et al. (2008) A new look at XXYY syndrome: medical and psychological features, American Journal of Medical Genetics – Part, vol.146, p. 1509-1522
– The 1000 Genomes Project Consortium (2015) A global reference for human genetic variation, Nature, vol.526, p. 68-74
– The Chimpanzee Sequencing and Analysis Consortium (2005), Initial sequence of the chimpanzee genome and comparison with the human genome, Nature, vol.437, p. 69-87
– The International HapMap Consortium (2007) A second generation human haplotype map of over 3.1 million SNPs, Nature, vol.449, p. 851-861
– TUKIAINEN Taru, […], MACARTHUR Daniel G. (2017) Landscape of X chromosome inactivation across human tissues, Nature, vol.550, p. 244-248
– VARKI Ajit, ALTHEIDE Tasha K. (2005) Comparing the human and chimpanzee genomes: searching for needles in a haystack, Genome Research, vol.15, p. 1746-1758)
– VARKI Ajit, NELSON David L (2007) Genomic comparisons of humans and chimpanzees, Annual Review of Anthropology, vol.36, p. 191-209
– YOSHIDA K., SUGANO S. (1999) Identification of a novel protocadherin gene (PCDH11) on the human XY homology region in Xq21. 3, Genomics, vol. 62, p. 540-543.
– ZHANG Yuchao et al. (13 nov 2015) Suppementary table S3 publiée en ligne sur https://academic.oup.com/mbe/article/33/1/302/2579282, erratum de Zhang et al. (2013) Genes that escape x-inactivation in humans have high intraspecific variability in expression, are associated with mental impairment but are not slow evolving, Molecular Biology and Evolution, vol.30(12), p. 2588-2601
– ZARREI Mehdi et al. (2015) A copy number variation map of the human genome, Nature Reviews Genetics, vol.16, p.172-183
____________________________
Notes
[1] Informations identiques à celles figurant dans son manuel (SVT 3è, Belin, 2008) hormis le nombre de gènes, dont le manuel indique qu’il est compris entre 20 000 et 30 000.
[2] Selon Jangravi et al. (2013), les gènes codants des familles TSPY (6 selon HCGN 11/2017), CDY (4 id.), BPY2 (3 id.) et VCY (2 id.) ne s’expriment que dans les testicules, ceux des familles RBMY (6 id.) et DAZ (4 id.) s’expriment « exclusivement dans les cellules germinales » (p. 13), ceux de la famille HSFY (2 id.) principalement dans les cellules germinales » (et uniquement dans les testicules selon Bellott et al. 2014), et ceux de la famille PRY (2 id.) « dans certains spermatozoïdes et spermatides » (p. 11). A noter que toujours selon Jangravi et al. (2013), les gènes de la famille XKRY (dont on ne sait toujours pas s’ils sont codants selon HCGN 11/2017) ne s’expriment également que dans les testicules.
[3] Les deux seuls exemples sur lesquels je suis tombée sont l’article de Skaletsky et al. (2003), consacré à la région MSY du Y humain, et Powers et al. (2017) qui est un plaidoyer pour l’analyse systématique des différences entre les sexes dans les études de génétique. Le premier se termine par un paragraphe intitulé « Genetic and biological differences between males and females » dans lequel on peut lire ceci : « It is commonly stated that the genomes of two randomly selected members of our species exhibit 99.9% nucleotide identity. In reality, this statement holds only if one is comparing two males, or two females. If one compares a female with a male, the second X chromosome (160 Mb, or roughly 3% of the diploid DNA content) is replaced by the largely dissimilar Y chromosome (60 Mb, or 1% of the diploid DNA content). This common substitution of the Y chromosome for the second X chromosome dwarfs all other DNA polymorphism in the human genome ». Dans Powers et al. (2017) on peut lire ceci : « However, the copy of the X-chromosome that is inactivated differs within each cell and some genes (~15%) on the second X-chromosome avoid X-inactivation altogether, which can lead to increased expression of the X-linked gene(s) in females compared to males [15]. […] To put this in perspective, unrelated human males share 99.9% of their genomes while unrelated males and females share only about 98.5% [16] (although this estimate is somewhat controversial). This could mean that the genome of a human male may be more similar to the genome of a male chimpanzee, sharing around 98.8% […] ». Significativement, la taux de ressemblance retenu entre homme et chimpanzé mâle est un vieux chiffre incorrect, la référence 15 est Carrel & Willard (2005), et la référence 16 est fantaisiste car il s’agit de Trabzuni et al. (2013), qui ne contient pas cette estimation.
[4] L’inactivation d’un X sur deux dans les cellules XX touche alétoirement dans chacune soit le X hérité du géniteur, soit celui hérité de la génitrice. De ce fait, si une mutation délétère a été héritée de l’un des deux, la maladie induite chez une personne XX pourra être moins grave que chez une personne XY ayant hérité du X muté, l’expression du X non muté dans une cellule sur deux en moyenne pouvant plus ou moins compenser chez elle le défaut du X muté.
[5] Selon Ensembl v90 (2017), dont les données sur le chimpanzé les plus récentes datent de 2012, la longueur totale de son génome est d’environ 3146 Mb (2963 Mb sur les autosomes, 157 Mb sur le X, 26 Mb sur le Y et moins de 17 kb dans l’ADN mitochondrial), mais Prüfer et al. (2012) n’ont pu en aligner qu’environ 2720 Mb sur le génome humain. Leurs informations supplémentaires en ligne soulignent que le séquençage du génome du chimpanzé n’est pas terminé, notamment pour le chromosome X. Dans Ensembl v90 (2017), seuls 18 282 gènes codants sont décomptés (17 530 sur les autosomes, 649 sur le X, 90 sur le Y et 13 dans l’ADN mitochondrial), ce qui correspond à près de 10 % de moins que dans le génome humain. En outre, presque trois fois moins de gènes non codants et de pseudogènes que dans le génome humain sont actuellement répertoriés dans cette base.
[6] Deux gènes homologues sont dits orthologues si on pense que le gène ancestral dont ils sont issus était présent chez le dernier ancêtre commun, et paralogues si on pense qu’ils dérivent d’une duplication du gène ancestral.
[7] cf Reich (2001), p.128-129 : « Conformément aux prévisions antérieures, il est désormais fermement établi que l’ensemble de l’espèce humaine dispose d’un génome identique à 99,9%, tandis que la variation entre les membres de l’espèce n’est que de 0,1 %. […] les études pilotes ainsi que les évaluations expérimentales sur les ressemblances globales ont révélé que le génome du chimpanzé, lorsqu’il est compté en séquence texte, est identique à environ 98,5% à celui de l’Homo sapiens. […]. La différence de 1,5% est étonnante, car la différence entre humains eux-mêmes peut être beaucoup plus importante en termes de pourcentage lorsque nous comparons les femmes et les hommes, étant donné que le chromosome X (les femmes en ont deux et les hommes un seul, le chromosome Y remplaçant le second chromosome X) contient environ 5% des informations de notre génome, ce qui pourrait signifier que les hommes et les femmes sont différents à 5%, c’est-à-dire davantage que les humains et les chimpanzés du même sexe.»
[8] Sur Serge Ginger, ses sources et des extraits de son discours relayé dans divers médias, voir https://allodoxia.odilefillod.fr/2015/08/02/les-pouvoirs-extraordinaires-de-france-2-part1/#note8, https://allodoxia.odilefillod.fr/2015/08/02/les-pouvoirs-extraordinaires-de-france-2-part1/#note9, https://allodoxia.odilefillod.fr/2015/08/02/les-pouvoirs-extraordinaires-de-france-2-part1/#note16 et https://allodoxia.odilefillod.fr/2015/08/02/les-pouvoirs-extraordinaires-de-france-2-part1/#note20 .
[9] Voir sa page www.psycho-ressources.com/bibli/femmes-et-hommes.html mise en ligne en 2003, reprenant le texte d’une conférence donnée à maintes reprises, notamment en octobre 2001 dans le cadre d’un congrès de la European Association for Psychotherapy et au 3ème Congrès mondial de psychothérapie en 2002, et dont il explique qu’il la développe habituellement sous la forme d’un séminaire de 4 journées pleines pour ses étudiants. Sur ses sources, voir https://allodoxia.odilefillod.fr/2015/08/02/les-pouvoirs-extraordinaires-de-france-2-part1/#note9.
[10] Selon le site de l’éditeur, Michel Badoc, professeur émérite à HEC-Paris, donne « des cours et conférences relatives aux neurosciences et marketing, marketing sensoriel… » et est conseiller auprès de sociétés internationales ; Anne-Sophie Bayle-Tourtoulou, professeure associée à HEC-Paris, donne des cours et conférences « en marketing, neurosciences et marketing, marketing sensoriel, distribution ».
[11] En 1999, Carrel, Willard et leurs collègues rapportent les résultats d’une étude de l’expression de 224 « gènes » (connus ou putatifs, et codants avérés ou présumés codants) du chromosome X humain dans des cultures de cellules hybrides rongeur/humain (Carrel et al. 1999). Le groupe a trouvé que 34 sur 224, soit 15%, échappent à l’inactivation dans toutes les lignées cellulaires de leur échantillon, et que 13 autres (soit 6%) échappent à l’inactivation dans certaines d’entre elles. Les auteur·es ont également testé l’expression de 10 de ces gènes dans des cultures de fibroblastes humains, et indiquent qu’elle a donné des résultats « complètement cohérents ».
[12] J’ai eu du mal à trouver ces informations car Carrel & Willard (2005) ne disent rien sur les cellules hybrides rongeurs-humains utilisées, renvoyant pour leur description à Carrel et al. (1999), or lui-même renvoie à Brown et al. (1997), et ce dernier renvoie lui-même à deux études plus anciennes que j’ai renconcé à aller consulter. Brown et al. (1997) donne déjà deux informations : les rongeurs en question sont des souris, et il est indiqué que la portée de l’analyse de ces hybrides est limitée par le fait qu’elle ne permet d’étudier que les gènes s’exprimant dans les fibroblastes. Par ailleurs, dans le communiqué de presse émis par Penn State, Carrel indique que les cellules humaines utilisées dans l’étude sont des cellules de la peau, et Lyon (2005) indique de son côté que les cellules hybrides étaient des « cultured skin fibroblasts ».
[13] C’est ce qui est indiqué dans le texte de l’article. Toutefois, la Table S1 en ligne à laquelle il renvoie (qui liste ces gènes, mais sans rapporter le résultat du test pour chacun malheureusement) en contient 100, et selon la Table S4 en ligne seuls 93 gènes ont pu être testés à la fois dans les fibroblastes humains et les hybrides.
[14] On retrouve ce résultat dans la Table s4 en ligne. A noter que l’examen de cette table indique aussi que sur 40 gènes inactivés dans toutes les lignées hybrides et aussi testés dans les fibroblastes humains, 3 échappaient à l’inactivation dans « certains » échantillons de ceux-ci. On ne peut savoir lesquels, car encore une fois les données détaillées par gène ne sont pas disponibles concernant l’expression dans les fibroblastes humains.
[15] A noter que Julianna Kettlewell, journaliste scientifique, a fait un bref compte rendu de l’étude dès le soir du 16/03/2005 sur le site de BBC News, correct au moins dans la mesure où elle laisse dans leur bouche les propos des scientifiques qu’elle a interviewés pour la décrire/commenter. Son article pêche (afflige) toutefois par sa conclusion, dans laquelle elle se base sur un article de New Scientist pour affirmer ceci : « les hommes ont plus de chance d’avoir un retard mental, mais ils ont aussi plus de chances d’être des génies. Bien que le QI moyen des hommes et celui des femmes soient égaux, on trouve plus souvent des hommes aux deux extrémités de l’intelligence. C’est parce que si vous avez de très bons gènes de l’intelligence sur votre chromosome X, il est avantageux qu’ils ne soient pas étouffés par des gènes plus moyens portés par un autre chromosome X » (ma traduction). Si on sait bien expliquer pourquoi le second X rend effectivement les femmes moins sujettes à certaines formes de retard mental, le reste n’est en revanche que pure spéculation.
[16] On ne comprend pas bien comme Holtz (ou Willard) a pu arriver à 2 %, car c’est la toute nouvelle estimation de 1098 gènes sur le X qui est retenue dans l’article, et il est par ailleurs indiqué qu’on estime qu’il y a 30 000 gènes en tout. Même en estimant, comme c’est fait dans l’article, que 25 % des gènes du X sont exprimés différemment chez les femmes (au diable les nuances !), soit 300 gènes après arrondi (bis !), même en ajoutant le nombre estimé par Ross et al. (2005) de 78 de gènes sur le Y, et même en commettant la même erreur grossière que celle que j’ai soulignée dans le livre de Mattei (consistant à oublier de doubler le numérateur pour prendre en compte le génome diploïde), le compte n’y est pas (478 / 30 000 = 1.25 %).
[17] Steve Connor attribue un comportement au X, identifie le X aux femmes et le Y aux hommes, et invente une lutte entre les deux chromosomes reflétant sa vision de la « guerre des sexes ». Maureen Dowd personnifie quant à elle les gènes du X et tente d’inverser la hiérarchie sociale des sexes : alors qu’on les pensait « dociles et inertes, se prélassant sur une méridienne évolutive victorienne », les gènes du second X « sont actifs, conférant aux femmes une expression des gènes significativement supérieure à celle des hommes ». Sous sa plume, les femmes sont ainsi « plus complexes », les hommes sont privés d’un X supplémentaire remplacé par un pauvre Y dégénéré, et le titre de son article est un jeu de mot sur l’expression « plafond de verre » (ce qui empêche les femmes d’accéder aux plus hauts niveaux de revenus et de pouvoir) suggérant que ce sont en fait les hommes qui sont coincés sous un « plafond cellulaire » du fait de leur manque d’un second X. Pour une réflexion sur la promotion du chromosome X comme symbole de la féminité auquel les femmes sont censées s’identifier et dont elles sont censées être fières, voir Richardson (2012).
[18] Par exemple la journaliste de Sciences et Avenir prétend que « la moitié des gènes actifs découverts par les chercheurs américains correspondent à des gènes également présents sur le chromosome Y », et que ces gènes « sont donc doublement exprimés chez les deux sexes » (do O’Gomes 17/03/2005).
[19] Par exemple, le « journaliste » de RTFlash fait dire à Willard que la variabilité de l’activité des gènes du X chez les femmes contraste avec leur activité relativement « stable » chez les hommes. Sciences et Avenir choisit de titrer « Les chromosomes X : des atouts considérables pour les femmes », et la journaliste explique : « Pour les femmes, cette mosaïque d’expressions possibles pour le chromosome X est un atout non négligeable. Elle lui offre plus de flexibilité face aux mutations que son patrimoine génétique peut subir ». Simone Gilgenkrantz (2005) propose quant a elle dans Médecine/Sciences des métaphores se rapprochant par certains aspects de celles de Dowd. Ainsi, elle relate les résultats de l’étude en terme d’ « insubordination » du chromosome X, et souligne que le profil d’expression de chaque cellule XX « en devient imprévisible » : « Nous le savions déjà : la femme… ni tout à fait la même, ni tout à fait une autre… ». Elle parle par ailleurs de la « sollicitude » du chromosome X « vis-à-vis de la masculinité et de son compagnon, le chromosome Y », métaphore plus pertinente que celle de Steven Connor sur le plan biologique (loin de « se battre » contre le Y, le X s’est au contraire en quelque-sorte « adapté » à la dégénérescence du Y), mais qui n’en reste pas moins ancrée dans une certaine idéologie du genre.
[20] Je suppose que le billet est de sa plume puisqu’elle se permet d’en faire un vaste copié/collé sans en citer la source dans son livre Ex Utero. Pour en finir avec le féminisme (2009). Par ailleurs, ce billet daté du 22 mars 2005 (sans doute initialement publié sur le site www.hyperdarwin.net qu’elle alimentait avec Charles Muller) est publié sur www.lesmutants.com, site anonyme ayant remplacé www.hyperdarwin.net mais dont elle est déclarée créatrice et administratrice du contenu depuis 2006. Sur lesmutants.com, voir https://allodoxia.odilefillod.fr/2014/07/23/camion-poupee-jeux-singes/#note14, ainsi que la sympathique page www.lesmutants.com/luttedesclasses.htm contenant la profession de foi du site en sept points : « Point 1 – Du fait de la révolution technique et scientifique moderne, le mode de production des ressources et des richesses est devenu de plus en plus dépendant de la capacité cognitive générale des individus. Point 2 – Les hiérarchies socio-économiques entre individus et entre nations sont positivement corrélées à la mesure de la capacité cognitive générale (QI) des individus et des populations. Point 3 – La capacité cognitive générale étant en large partie héritable, un nombre croissant d’individus et de nations deviendront inadaptés en l’absence d’intervention sur le pool génique humain. Ils seront condamnés à une existence de parasites ou d’esclaves. Point 4 – La lutte contre l’exclusion est un leurre destiné à masquer une évidence : il est impossible d’inclure des individus ne possédant pas les qualités psychobiologiques requises par leur système de production. Point 5 – Le développement égal pour tous est un mythe destiné à masquer une évidence : il est impossible de développer des sociétés ne possédant pas les qualités psychobiologiques requises par le développement. Point 6 – L’islamisme est une illusion destinée à masquer une évidence : la plupart des peuples décolonisés ont été à ce jour incapables d’atteindre un niveau de richesse et de puissance comparable à celui de leurs anciens colonisateurs. Point 7 – Les points précédents sont connus, mais ne sont pas exprimés. Ce n’est pas seulement une affaire idéologique. Une telle désillusion nuirait à la bonne marche du commerce. Car dans consommateur, il y a con… »
[21] Nguyen & Disteche (2006) ont étudié le niveau d’expression des gènes du X dans un échantillon de cellules extraites de divers tissus (rate, foie, coeur, muscle squelettique, hypothalamus et autres regions cérébrales), et n’ont pas trouvé de différence significative entre femmes et hommes. En se focalisant sur les 27 gènes échappant le plus souvent à l’inactivation dans leurs échantillons et sur trois types de tissus humains, les auteur·es on trouvé que leur niveau moyen d’expression était supérieur de 11 % seulement dans les tissus provenant de femmes, ce qui s’expliquerait selon Nguyen et Disteche à la fois par le faible niveau moyen d’expression des gènes échappant à l’inactivation et par l’effet d’autres différences entre les sexes telles que les différences hormonales.
[22] ALG13, CA5B, DDX3X, EIFIAX, EIF2S3, FUNDC1, HDHD1A, JARID1C, MSL3L1, PCTK1, PNPLA4, PRKX, RPS4X, SMC1L1, STS, UBE1, USP9X, UTX, ZFX et ZRSR2. On retrouve en particulier le gène STS identifié dès 1979 comme échappant à l’inactivation au moins chez certaines femmes, ainsi que HDHD1A, PNPLA4, RPS4X, UTX et ZFX que Talebizadeh et al. (2006) avaient trouvé surexprimés chez les femmes dans au moins trois des tissus étudiés.
[23] Les 6 gènes au plus haut ratio F/H d’expression moyen, égal à « environ 1.5 » (+ 50 % d’expression chez les femmes), sont JARID1C, UTX, HDHD1A, PNPLA4, DDX3X et EIF1AX. Les auteur·es indiquent qu’a contrario, EIF2S3, USP9X, CA5B, PCTK1, ZFX et SMC1L1 présentent tous un ratio inférieur à 1.2 (+ 20 %).
[24] Sur 318 gènes du X ayant pu être testés dans 1 à 61 échantillons de lymphocytes immortalisés issus de plusieurs personnes, Zhang et al. (2015[2013]) n’en ont trouvé aucun échappant à l’inactivation dans tous les échantillons testés, à part deux n’ayant pu être testés que dans un seul échantillon (l’un de ces gènes n’avait pas été étudié par Carrel et Willard, et l’autre avait été jugé inactivé par eux). Ils ont trouvé 114 gènes échappant à l’inactivation dans au moins deux échantillons, mais 66 d’entre eux avaient été déclarés inactivés par Carrel & Willard (2005). Parmi les 264 gènes communs à ces deux études, Carrel & Willard (2005) avaient identifié 32 gènes « échappant à l’inactivation » et 52 y échappant parfois, mais sur ces 84 gènes, seuls 39 ont été jugés échapper à l’inactivation dans plus d’un échantillon de Zhang et collègues. Parmi les 200 gènes communs à la fois à Zhang et al. (2013), Carrel & Willard (2005) et une troisième étude sur la même question (Park et al. 2010), Zhang et collègues ne trouvent que 7 gènes ayant été jugés « échapper à l’inactivation » par les deux autres équipes et y échappant dans plus de 50 % de leurs échantillons.
[25] Cotton et al. (2013) ont considéré qu’un gène était inactivé sur Xi si son niveau d’expression était inférieur à 10 % de son niveau d’expression sur Xa. Ensuite, un gène a été considéré comme « sujet à l’inactivation » s’il était inactivé chez au moins 78 % des femmes de leur échantillon pour lesquelles les données étaient exploitables, comme « échappant à l’inactivation » s’il était activé chez au moins de 78 % d’entre elles, et « à échappement variable » dans les cas intermédiaires. Sur 409 gènes (incluant des gènes non codants) examinés, les auteur·es ont trouvé 68 gènes « échappant » dont 35 dans PAR1 (ce qui est étrange car selon Ensembl v90 [2017], les régions PAR contiennent moins de 30 gènes), c’est-à-dire seulement 33 gènes hors PAR1, soit environ 9 % des gènes hors PAR1 testés. 146 à 148 gènes selon les paragraphes de l’article (soit 29 % du total) ont été classés à « échappement variable ». En moyenne, le niveau d’expression sur Xi par rapport à celui sur Xa était de 37 % pour les 33 gènes hors PAR1 « échappant », 18.9 % pour les 147 gènes « variables », et 5.3 % pour les 295 gènes « sujets à l’inactivation ». Rem : pour PAR2, les auteur·es trouvent que SPRY3 est « variable » et de niveau d’expression sur Xi égal à 10.4 % de celui sur Xa, que SYBL1/VAMP7 échappe toujours et de niveau d’expression sur Xi égal à 0.61 %, et « étonemment » que IL9R est « sujet à l’inactivation » (contrairement aux études précédentes). Les auteur·es signalent que des données suggèrent que les gènes SPRY3 et SYBL1/VAMP7 du chromosome Y ne s’expriment pas chez les hommes.
[26] « C’est ainsi qu’en 2005, Huntington Willard, un des 250 co-auteurs de l’annotation du chromosome X, déclare dans Nature : “Il n’y a pas un génome humain, mais bien deux : le génome mâle et le génome femelle“. […] Dans leur étude, Willard et Carrel confirment qu’environ 15 % des gènes du second chromosome X restent actifs, à des degrés divers et dans des régions très différentes du chromosome. Plus étonnant encore, une autre proportion importante de gènes (environ 10 %) s’exprime très différemment selon les femmes. Cette hétérogénéité féminine est totalement absente chez l’homme. Au total, ce sont ainsi 200 à 300 gènes (25 % du chromosome X) dont l’expression varie selon les sexes, présente chez l’un, absente chez l’autre. » (Sastre 2009)
[27] Dans https://allodoxia.odilefillod.fr/2015/08/06/les-pouvoirs-extraordinaires-de-france-2-part2/#note32 et https://allodoxia.odilefillod.fr/2015/08/06/les-pouvoirs-extraordinaires-de-france-2-part2/#note33.
[28] On notera l’habile inclusion de cette précision afin de donner du poids à la phrase qui va suivre, d’autant que comme dans Ex utero en 2009, Peggy Sastre cite ici en référence l’article de Ross et al. dans Nature.
[29] Extrait de Junien (2011), paragraphe consacré au « dimorphisme sexuel des marques épigénétiques » : « Il existe d’importantes différences entre les sexes non seulement en termes de comportements et de préférences alimentaires, de réponses aux nutriments et aux médicaments et d’apparition ou de fréquence de certaines maladies mais aussi en termes de programmation au cours du développement comme le révèlent de plus en plus d’études chez toutes les espèces étudiées. Les fœtus et les placentas mâles et femelles ne réagissent pas nécessairement de la même façon à des stimuli environnementaux, lors de phases précises du développement, et les conséquences à long terme diffèrent également [3, 43]. Le dimorphisme sexuel repose aussi sur des différences épigénétiques qui affectent l’expression différentielle d’une proportion importante de gènes (10 à 30 %) dans tous les tissus [44] en liaison avec les chromosomes sexuels [45], les hormones et les influences socio-culturelles. Ces différences, très précoces pour certaines, bien avant la différenciation des gonades [46], sont encore mal explorées mais devraient faire l’objet d’une attention plus soutenue, tant les stratégies thérapeutiques ou préventives qui valent pour un sexe ne sont pas nécessairement adaptées à l’autre sexe [3, 47-49]. » La référence 3 est la revue de la littérature déjà citée dont elle est co-auteure (Gabory et al. 2009), les références 43 et 44 sont des études menées sur des souris et la référence 46 une étude mené sur des embryons de bovins, et la référence 45 est Davies et Wilkinson (2006), revue de la littérature suggérant des explications de la sexuation du cerveau complémentaires ou alternatives aux hormones. Les références 47 à 49 sont McCarthy et al. (2009) The epigenetics of sex differences in the brain, Jour.of Neurosci. Vol.29 ; Penisi E. (2009) Sex and social structure, Science, vol. 326 ; Shepard et al. (2009) Genetic, epigenetic and environmental impact on sex differences in social behavior, Physiology & Behavior vol. 97.
[30] Extrait de Scheen & Junien (2012), paragraphe consacré au « dimorphisme sexuel des marques épigénétiques » : « Il existe d’importantes différences entre les sexes en termes d’apparition ou de fréquence de certaines maladies complexes, phénomène bien connu dans l’espèce humaine (22). Ces différences liées au sexe existent également en termes de programmation au cours du développement, comme le révèlent les études réalisées dans différentes espèces animales. Les fœtus et les placentas mâles et femelles ne réagissent pas nécessairement de la même façon à des stimuli environnementaux, lors de phases précises du développement, et les conséquences à long terme diffèrent également. Le dimorphisme sexuel repose aussi sur des différences épigénétiques qui affectent l’expression différentielle d’une proportion importante de gènes (10 à 30 %) dans tous les tissus en liaison avec les chromosomes sexuels, les hormones et les influences socio-culturelles (23). Ces différences, très précoces pour certaines, bien avant la différenciation des gonades, sont encore mal explorées dans l’espèce humaine. Elles devraient, cependant, faire l’objet d’une attention plus soutenue, tant les stratégies thérapeutiques ou préventives qui valent pour un sexe ne sont pas nécessairement adaptées à l’autre sexe (23). » La référence 22 est Kaminsky et al. (2006) Complex disease, gender and epigenetics, et la référence 23 est Junien et al. (2012) déjà cité (« Le dimorphisme sexuel au XXIe siècle ». Scheen & Junien (2012) est cité en référence dans un rapport du sénat publié en 2016 dont elle a participé au pilotage, à l’appui du paragraphe suivant : « [Le dimorphisme sexuel] a trait aux importantes différences existant entre les sexes en termes d’apparition ou de fréquences de certaines maladies complexes, selon la définition donnée par Mme Claudine Junien. Ces différences liées au sexe existent également en termes de programmation au cours du développement, comme le révèlent les études réalisées dans différentes espèces animales. Ces différences tiennent au fait que les foetus et les placentas mâles et femelles ne réagissent pas nécessairement de la même façon à des stimuli environnementaux, lors de phases précises du développement. Les conséquences à long terme diffèrent également. Le dimorphisme sexuel repose aussi sur des différences épigénétiques qui affectent l’expression différentielle d’une proportion importante de gènes (10 % à 30 %) dans tous les tissus en liaison avec les chromosomes sexuels, les hormones et les influences socio-culturelles. Ces différences, très précoces pour certaines, bien avant la différenciation des gonades, sont encore mal explorées dans l’espèce humaine. Elles devraient cependant faire l’objet d’une attention plus soutenue tant les stratégies thérapeutiques ou préventives qui valent pour un sexe ne sont pas nécessairement adaptées à l’autre sexe. » (www.senat.fr/rap/r16-033-1/r16-033-1.html).
[31] Lors de la séance de questions qui suit son exposé, un participant lui demande quelle est son analyse des problèmes d’ « incompatibilité entre le sexe et le genre » chez les personnes « transsexuelles » (24’30’’). Je suppose qu’il souhaitait savoir comment elle expliquait la « transsexualité », mais elle n’a pas compris la question ainsi. Voici ce qu’elle répond : « le problème auquel il faut penser, et auquel on ne pense pas suffisamment, c’est que le cerveau, in utero, se forme différemment selon la composition chromosomique, donc si vous avez une composition XY ou XX, la formation du cerveau va être différente, et à cela va s’ajouter aussi les hormones. Donc quand on fait un changement de sexe, […], on ne va pas forcément tenir compte des différences qui sont déjà mises en place dans le cerveau, et il peut y avoir là aussi, effectivement, incompatibilité entre le sexe tel qu’il va être visible et puis les différences au niveau du cerveau… […] »
[32] Les éléments (et diapositives) clés de sa conférence sont repris dans un document pdf qui a été un temps disponible en ligne (téléchargé par moi le 17 mai 2016), finalisé le 17 janvier 2016, créé par « Claudine Junien 1 » à partir d’un document Word intitulé « PLaidoyer DIMORPHISME SEXUEL FIGURES C JUNIEN 011215.doc » (titré « Les différences biologiques entre l’homme et la femme sont sous-estimées en Recherche et en Médecine », « Pr Claudine Junien – Paris le 1er Décembre 2015 »). On peut notamment y lire ceci (en gras dans le texte) : « Dès l’instant de notre conception, chacun d’entre nous est doté d’un sexe biologique. Sur les 23 paires de chromosomes héritées de nos parents, c’est la paire de chromosomes sexuels qui fait toute la différence : soit 2 chromosomes X (XX) pour une fille, soit un chromosome X et un chromosome Y (XY) pour un garçon. Il n’existe que de très rares cas (0,018 % des naissances) pour lesquels le sexe, à la naissance, peut être ambigu, que l’origine soit chromosomique, génique, développementale ou environnementale. […] Mais, en fait, chez la fille, 15 à 25% des 1400 gènes de l’X échappent partiellement à cette inactivation et ont donc un taux d’expression supérieur à celui des garçons. De plus, les gènes spécifiques du chromosome Y s’expriment exclusivement chez le mâle. Ainsi, si la ressemblance génétique, en termes de séquence, entre 2 hommes ou 2 femmes est de 99,9%, la ressemblance entre un homme et une femme n’est que de 98,5% ; soit le même ordre de grandeur qu’entre un humain et un chimpanzé, de même sexe. […] Le génome dont nous héritons de nos parents est stable et définitif. Alors comment expliquer que nos 23 000 gènes ne s’expriment pas de la même façon dans le foie, le rein ou le cerveau et avec des différences selon le sexe ou l’age ? […] Enfin, avoir peur de ces différences, ou les nier par des moyens détournés, est absurde et contre-productif. Les levées de bouclier contre des médicaments qui traitent mieux ou moins bien les patients selon qu’ils sont de telle ou telle origine ethnique ou de tel ou tel sexe – un anti‐hypertenseur qui a un degré d’efficacité supérieur selon que le malade est noir ou blanc, ou le zolpidem (Stilnox) qui a déclenché l’ire de féministes américaines car les doses recommandées n’étaient pas identiques pour les hommes et les femmes – sous prétexte que ce serait de la discrimination – oublient qu’au final, sans cette « discrimination », des patients sont moins bien soignés justement à cause de leur sexe ou de leur race… »
[33] « De fait, si la société multiplie les préjugés et les idées fausses sur les théories du genre, la médecine, elle, a au contraire tendance à lisser des différences pourtant bien réelles entre les hommes et les femmes. C’est particulièrement le cas en France, souligne l’Académie de Médecine, qui appelle à s’inspirer des approches américaines, allemandes ou encore suédoises, davantage centrées sur la médecine différenciée. “Si on compare les séquences d’ADN, on s’aperçoit qu’il y a 1 % de différences entre deux hommes ou deux femmes, explique la généticienne Claudine Junien, à l’origine de ce rapport. En revanche, parce que la femme a deux chromosomes X et l’homme, un chromosome X et un Y en plus, on se retrouve avec 15 fois plus de différences !” En fait, il y aurait autant de différences sur le plan de l’ADN entre un homme et une femme qu’entre un humain et un chimpanzé ! Ces disparités ont de nombreuses conséquences. Comme le montre le rapport de l’Académie de Médecine, certaines maladies touchent plutôt les femmes (Alzheimer, dépression, ostéoporose, sclérose en plaques…) quand d’autres atteignent davantage les hommes (autisme, tumeurs du cerveau, AVC ischémique…). »
[34] « J’ai appris plein de choses cette semaine grâce à l’Académie de Médecine, qui lançait un appel pour “une vraie parité en santé” entre hommes et femmes – pour une fois d’ailleurs, la parité était aussi sur l’estrade, alors que d’ordinaire le ratio de cette auguste assemblée est plutôt d’une (femme) pour dix (hommes)… Ceci d’abord : sur le plan génétique, il y a 99,9% de similitude entre deux hommes (ou deux femmes) pris au hasard. En revanche, il n’y a “que” 98,5% de similitude entre un homme et une femme. Or curieusement, 98,5%, c’est aussi le pourcentage de similitude entre un homme et un singe mâle (ou entre une femme et un singe femelle). Je ne sais pourquoi, il m’est venu alors cette idée saugrenue que, génétiquement, François Hollande est aussi éloigné de Ségolène Royal que d’un chimpanzé mâle. »
[35] « Mieux encore, et hautement plus dérangeant, ils [« les académiciens tricolores »] estiment que la vision actuelle sur la différenciation du sexe est déjà devenue obsolète. […] En effet, si la ressemblance, en termes de séquence, entre deux hommes ou deux femmes est de 99,9 %, la ressemblance entre un homme et une femme n’est que de 98,5 % – du même ordre de grandeur qu’entre un humain et un chimpanzé de même sexe… »
[36] « Si la ressemblance génétique en termes de séquence entre deux hommes ou deux femmes est de 99,9 %, la ressemblance entre un homme et une femme n’est que de 98,5 %. Or “c’est le même ordre de grandeur qu’entre un humain et un chimpanzé de même sexe”, insiste-t-elle [Claudine Junien]. » A noter que les journalistes laissent prudemment dans le bouche de Claudine Junien la comparaison avec le chimpanzé, mais reprennent à leur compte l’idée qu’il y a 1.5% de différence entre femmes et hommes.
[37] « Titre insolite d’un article de Le Figaro (02/09/2016) : Médecine : les différences hommes-femmes négligées. La France est très en retard dans ce domaine, que ce soit dans la recherche ou dans le choix des traitements. Le lecteur s’imagine encore une propagande en faveur du féminisme. Il n’en n’est rien. Il est mis l’accent sur le fait qu’on ne soigne pas un homme comme une femme. L’Académie de Médecine vient de s’intéresser la question « pour faire bouger les choses ». La France est très en retard sur le sujet explique Claudine Junien, généticienne, Professeur à la Faculté de Paris-Ouest. « On se préoccupe beaucoup de parité surtout sur le genre, mais il faut aussi faire prendre conscience du sexe biologique » explique-t-elle. Voilà qui est intéressant et ne fait certainement pas les affaires de LGBT. La ressemblance entre un homme et une femme n’est que de 98,5 %. Or « c’est le même ordre de grandeur qu’entre un humain et un chimpanzé de même sexe ». En d’autres mots, il y a autant de différences entre un homme et une femme qu’entre un humain et un chimpanzé. […] »
[38] « Y para ello, la doctora Junien explica que “se sabe que, desde su concepción, el embrión masculino no se comporta de la misma manera que el embrión femenino” y eso que aún “las hormonas sexuales aún no han aparecido”. La genetista añade que la similitud genética entre dos hombres o dos mujeres es del 99,9% mientras que el que se da entre un hombre y una mujer es del 98,5%. » On noter que sur ces sites catholiques, la comparaison avec le chimpanzé, pas vraiment compatible avec l’anthropologie vaticane, est supprimée. Sur le site d’Alliance Vita, qui a relayé un compte-rendu de la note de l’Académie des sciences dès le 24 juin 2016, c’est carrément toute la partie génétique qui est supprimée (https://www.alliancevita.org/2016/06/academie-de-medecine-nier-les-differences-entre-les-sexes-au-niveau-medical-cree-des-injustices/ ; l’article renvoie à une chronique de Peggy Sastre de 2013 sur Le Plus de L’Obs pour aller plus loin).
[39] « Si la ressemblance, en termes de séquence d’ADN, entre 2 hommes ou 2 femmes est de 99,9 %, la ressemblance entre un homme et une femme n’est que de 98,5 %, du même ordre de grandeur qu’entre un humain et un chimpanzé, de même sexe… Ces DLS [différences liées au sexe] existent dans toutes nos cellules qu’il s’agisse du foie, du cerveau ou des organes reproducteurs.»
[40] Dans la vidéo, Claudine Junien explique que la différence entre XX et XY « aboutit à des différences génétiques qui sont 15 fois plus grandes entre un homme et une femme qu’entre deux hommes » (3’20’’). Dans le texte de l’article, la médecin qui l’ interviewée écrit : « Dès la conception, l’embryon femelle ne se comporte pas comme le mâle : parmi les 23 paires de chromosomes humains, l’une d’entre elles détermine le sexe, XY pour les hommes et XX pour les femmes. Une lettre qui pèse lourd : les différences génétiques sont 15 fois plus importantes entre un homme et une femme qu’entre 2 hommes. […] Au final, la ressemblance entre un homme et une femme n’est que de 98,5%, soit le même ordre de grandeur qu’entre un humain et un chimpanzé ! »
[41] [Chapeau de la tribune :] « Si, génétiquement, la ressemblance entre 2 hommes ou 2 femmes est de 99,9 %, elle n’est que de 98,5 % entre un homme et une femme, comme entre un humain et un chimpanzé de même sexe ! Pour le professeur Claudine Junien, il est urgent de prendre en compte la différence entre les sexes dans la médecine. » Texte de Claudine Junien : « Si, génétiquement, la ressemblance entre 2 hommes ou 2 femmes est de 99,9 %, elle n’est que de 98,5 % entre un homme et une femme, comme entre un humain et un chimpanzé de même sexe! Du fait de leurs chromosomes sexuels, toutes les cellules de l’embryon sont déterminées sexuellement: XX pour les filles, XY pour les garçons et elles gardent la mémoire de leur sexe. Chez la fille, dans chaque cellule, un des 2 X, paternel ou maternel, est inactivé au hasard ; mais 15 à 25 % des 1 400 gènes de l’X ne le sont pas et s’expriment donc davantage que chez les garçons qui n’ont qu’un seul X et qui manifestent leur différence par la petite centaine de gènes spécifiques de leur chromosome Y. Sachant que le génome est stable, définitif et identique dans chacune de nos cellules, seulement des différences dues au sexe peuvent donc expliquer que nos 23.000 gènes ne s’expriment pas de la même façon dans le foie, le rein ou le cerveau… Ainsi, en moyenne, 30 % des gènes s’expriment toujours différemment selon le sexe dans chacune des 60.000 milliards de nos cellules. »
[42] « Pour une recherche et une médecine sexuellement différenciées : des faits biologiques irréfutables » : « Sous prétexte de parité, on a trop longtemps évité en France de reconnaître les différences biologiques liées au sexe (DLS), entre les hommes et les femmes, au nom de l’égalité mais au mépris des évidences scientifiques […] Or, le sexe biologique a trop longtemps été nié dans notre pays au profit de la primauté du genre, au nom de l’égalité des sexes alors que, rappelons le, si la ressemblance, en termes de séquence d’ADN, entre deux hommes ou deux femmes est de 99,9 %, la ressemblance entre un homme et une femme n’est que de 98,5 %, du même ordre de grandeur qu’entre un humain et un chimpanzé, de même sexe…Le séquençage du génome humain a pu répertorier une petite centaine de gènes sur le chromosome Y qui s’expriment uniquement dans les cellules d’un mâle. Quant au chromosome X il contient environ 1 500 gènes. On a longtemps cru que l’un des 2 chromosomes X était complètement inactivé au hasard dans toutes les cellules d’une femme, mais en fait environ 15 % échappent à cette inactivation et sont donc plus exprimés dans des cellules XX que dans des cellules XY. […] »
[43] Cf dans Junien (04/02/2017) : « Simone de Beauvoir (1956) avec sa célèbre phrase “on ne naît pas femme, on le devient” se serait-elle fourvoyée, et avec elle, toutes celles et tous ceux qui ont enfourché ce dogme ? Hélas oui : avant même que le petit blastocyste (une centaine de cellules) ne s’implante dans l’utérus de la mère, on note déjà, à ce stade, que 30 % des gènes qui s’expriment montrent des différences significatives entre le mâle et la femelle. »
[44] « Génétiquement, la ressemblance moyenne entre deux hommes ou deux femmes atteint les 99,9%, mais elle n’est que de 98,5% entre un homme et une femme, soit 15 fois plus important qu’entre un humain et un chimpanzé de même sexe ! […] Chez la fille, en effet, un des deux X, d’origine paternelle ou maternelle, est inactivé au hasard dans chaque cellule, et sur les 1 400 gènes de cet X « inactif », 15% à 25% peuvent échapper à l’inactivation et donc s’exprimer davantage. Avec leur X unique, les garçons, en revanche, ne manifestent leur différence que par la petite centaine de gènes spécifiques de leur chromosome Y. Telle une mémoire sexuelle, ces gènes du chromosome X chez la fille et ceux du chromosome Y chez le garçon, modulent au bon moment, dans la bonne cellule, l’expression d’autres gènes, de sorte que, 30 % de nos gènes en moyenne s’expriment différemment selon notre sexe dans tous nos tissus et donc dans chacune de nos cellules. » Rem : outre que le pdf de cet article de Causeur est mis en ligne sur le site de l’Institut Diderot, l’article est signalé dans la Revue de presse du Comité Consultatif National d’Ethique (n°330, 1er au 19 avril 2017).
[45] « La femme n’est pas un homme comme un autre ! Le 04 avril 2017, deux femmes, professeur de Génétique médicale et docteur en philosophie des sciences, protestent : Les femmes payent le prix du déni des différences biologiques. Alors que depuis 2002, l’Organisation mondiale de la Santé (OMS) prône l’équité entre les sexes, trop peu d’études prennent en compte le fait que, si “génétiquement, la ressemblance moyenne entre deux hommes ou deux femmes atteint les 99,9%, mais elle n’est que de 98,5% entre un homme et une femme, soit 15 fois plus important qu’entre un humain et un chimpanzé de même sexe !” ».
[46] 39 701 pour les hommes XY, et 39 745 pour les femmes XX en gardant la logique utilisée pour le calcul du numérateur, i.e. en ne contant pas les gènes du second X qui ne s’expriment pas car ils n’échappent pas à l’inactivation, ce qui fait 40 479 – 841 + 107.
[47] Pour conforter cette estimation, j’ai aussi tenté d’exploiter les données d’études plus vastes et plus récentes, mais quasiment en vain. Par exemple, Balaton et al. (2015) présentent une synthèse des principales études s’étant penchées sur le processus d’échappement à l’inactivation du X pour tenter d’établir une liste des gènes du X pour lesquels il existe des données à peu cohérentes, mais outre que le fichier en ligne devant contenir cette liste est indisponible, aucune quantification du biais de sexe dans l’expression des gènes concernés n’est fournie. Il est juste indiqué que (seulement) 5 % de tous les gènes du X sont en moyenne sur-exprimés chez les femmes et 1.7 % au contraire sur-exprimés chez les hommes, ce qui est du moins à peu près cohérent avec Talebizadeh et al. (2006). Dans la très récente étude de Tukiainen et al. (2017) qui aboutit à l’estimation qu’environ 23 % des gènes du X échappent au moins parfois à l’inactivation (régions PAR comprises, et toujours avec des seuils arbitraires qui font que ce pourcentage n’a guère de signification concrète), les auteur·es ont aussi fait des comparaisons entre femmes et hommes. Après avoir sélectionné 82 gènes du X codants ou non codants (ARN longs) préalablement décrits comme échappant à l’inactivation et n’ayant pas un niveau d’expression très bas, et étudié leur expression dans 29 types de tissus issus de plus de 70 personnes, les auteur·es ont trouvé que 61 gènes avaient une différence d’expression statistiquement significative entre femmes et hommes dans au moins un des 29 tissus, mais ne quantifient pas cette différence. De même, leur table 13 en ligne liste 171 gènes considérés comme « échappant » ou « à échappement variable », indiquant que 62 sont sans biais F-H d’expression significatif, 62 avec une sur-expression chez les femmes, 30 avec au contraire une sur-expression chez les hommes et 17 avec un biais d’expression hétérogène, mais là encore sans quantifier ces biais d’expression. Leurs informations supplémentaires en ligne indiquent seulement que hors PAR, « l’ampleur de la différence homme-femme d’expression des gènes échappant à l’inactivation est légèrement réduite » par rapport à l’estimation du niveau d’expression sur Xi/ Xa, la médiane de cette différence étant « par exemple » de 14 % dans la région Xp versus 21 % d’expression de Xi/Xa. Quoi qu’il en soit, en considérant que 23 % des gènes régions PAR comprises échappent à l’inactivation, soit 174 gènes hors PAR, et que leur différence moyenne d’expression entre femmes et hommes et au plus de 20 %, cela donne au final 35 « équivalents gènes » au plus, ce qui est cohérent avec mon estimation de 42 au plus.
[48] Lorsqu’on compte le nombre total de gènes, un gène dont plusieurs copies existent sur un même chromosome (gène dupliqué) n’est compté qu’une seule fois, or voir par exemple dans https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-016-2392-0 : « The retention of duplicated genes creates a stoichiometric disparity in the protein-protein interaction network, with the duplicated genes producing more proteins than the non-duplicated ones [35, 36, 37]. » J’ai cherché en vain une estimation consensuelle du nombre moyen de gènes duplications comprises dans un génome humain diploïde, et ne sais donc pas dans quelle mesure la prise en compte de cet aspect affecterait le calcul. Pour un exemple de gène, voir le cas de AMY1 codant pour l’amylase salivaire, dont jusqu’à 20 copies peuvent être trouvées dans un génome diploïde et pour lequel Perry et al. (2007) trouvent une corrélation positive entre nombre de copies possédées et quantité produite d’amylase salivaire.
[49] Par exemple lors de sa conférence de 2015 à l’Académie des sciences. NB : c’est un pourcentage qui est régulièrement cité dans la littérature scientifique, mais je n’ai pas vérifié la pertinence ni la solidité de cette estimation.

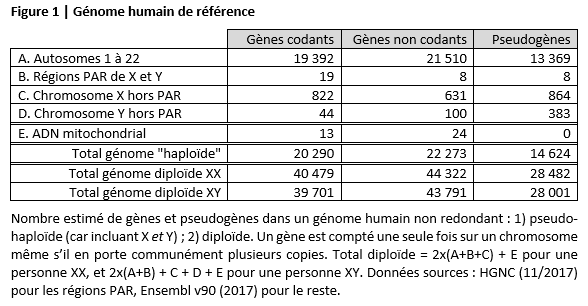
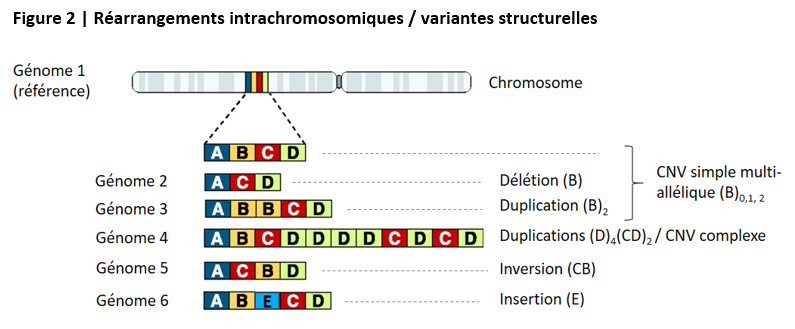
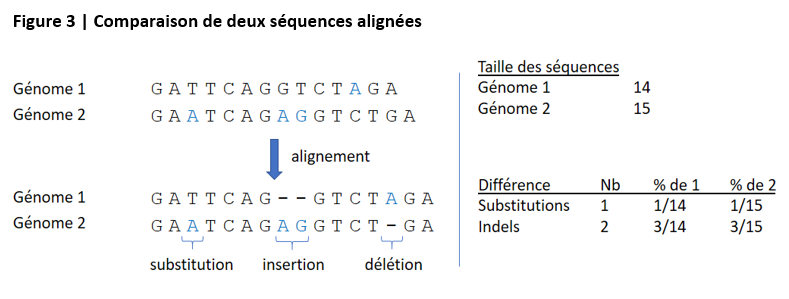

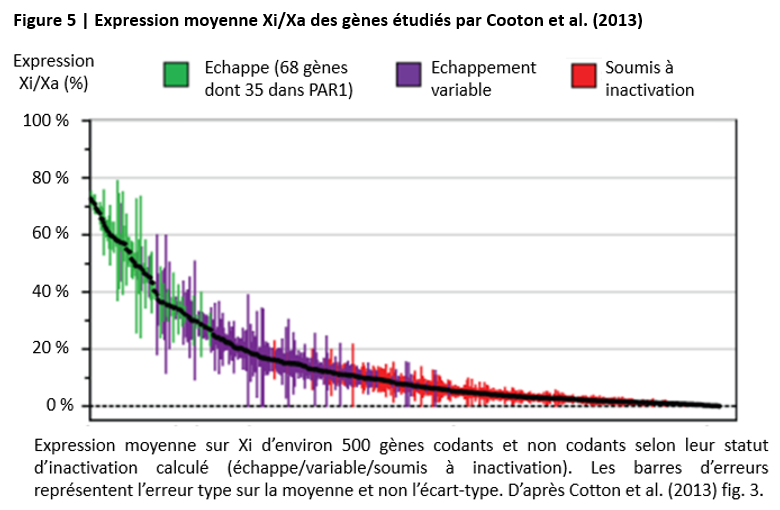
Bonjour, et merci pour cet article très intéressant.
Petite remarque: je vois que tout au long de l’article vous utilisez “mb” ce qui n’est je crois pas le prefixe SI correct (m étant “milli”). Je pense que “Mb” (pour mega) serait plus exact.
Merci. En effet, c’est une erreur de ma part. Je corrigerai dès que possible (pour l’instant il y a un souci technique qui fait que si je touche à l’article, il risque de passer en “privé”, donc je ne bouge pas).
Je pense que les chiffres précis ne sont pas tellement importants au regard de la conclusion qui est qu’une différence faible en nombre de gènes, même 0.2%, peut avoir des conséquences importantes quand on voit les différences humain.e.s et chimpanzés malgré tant de ressemblance au niveau du génome. Parce qu’évidemment, tout le monde avait bien remarqué que les hommes et les femmes sont plus sembables que les hommes et les chimpazés mâles…
Mais surtout en matière de différence génétique entre les hommes et les femmes, en tant que populations et non en tant qu’individus, il me semble que la différence essentielle est celle de la variabilité, moindre chez les femmes à cause de la lyonisation. Ce qui expliquerait des surreprésentations d’hommes dans les queues de distributions.
En effet ces chiffres ne sont pas importants, d’où l’inanité de l’argumentaire de Claudine Junien. Je voulais néanmoins souligner qu’en plus de mobiliser des calculs et comparaisons absurdes, elle avançait des chiffres faux.
Sur la deuxième partie de votre commentaire, je crois qu’on ne peut pas tirer de conclusion si hâtive. Carrel, Willard et les journalistes anglo-saxons qui ont relayé leur étude ont au contraire argué du processus d’échappement variable à la lyonisation pour prétendre que les femmes étaient plus variables que les hommes… Ce qui est certain, c’est que la lyonisation réduit en moyenne l’impact des mutations délétères portées par le chromosome X, et que ça explique sans doute au moins en partie la surreprésentation des hommes dans les troubles neuro-développementaux et le retard mental. Pour le reste, je demande à voir.
Merci beaucoup pour ce travail gigantesque. J’ai plusieurs fois entendu ce mythe absurde…
Édifiant.
Votre article est superbe, votre démonstration lumineuse.
Ça donne envie de douter (encore plus) de tout ce qui peut être dit ou écrit, par qui que ce soit, où que ce soit.
Bonjour Madame ! Merci infiniment pour tout ce travail, que je redécouvre avec joie à l’instant ou je suis empêtré dans des débats sans fins avec des amis qui virent masculinistes et se gavent de vidéos de Jordan Peterson (d’ailleurs, si un jour vous avez le temps / l’envie / le courage de dire un mot à son propos, cela m’aiderait surement !)
Sinon, je voulais juste vous dire qu’en SVT, au lycée, on évite au maximum le terme “code” pour parler du génome ou du patrimoine génétique, de peur que les élèves confondent avec le bon gros code génétique “historique”, c’est à dire le tableau de correspondance triplet de nucléotides –> acide aminé. C’est pourquoi je suis surpris de vous voir utiliser le terme ! (du coup vous l’aurez compris j’enseigne les SVT au lycée, et vous remercie au passage pour tout ce que vous faites à propos du clitoris)
Bonjour, merci pour votre commentaire. Je comprends votre remarque mais d’une part je parle à un moment de l’épissage alternatif, ce qui met à mal l’idée de correspondance univoque, et d’autre part (et surtout) le verbe “coder” et ses dérivés est vraiment un terme consacré dans ce domaine, et je dois dire que je ne vois pas trop comment l’exprimer autrement. Est-ce que vous avez une suggestion à me faire pour reformuler les nombreuses phrases dans lesquelles j’ai utilisé les termes code/codé(e)(s)/coder/codant ?
Ah ! En réalité, je pensais plus à son utilisation en tant que nom et pas en tant que verbe, par exemple dans ce passage :
” De ce fait, l’information génétique portée par l’ADN peut être vue comme un code écrit à l’aide de ces quatre lettres. La longueur d’un morceau d’ADN est exprimée en nombre de bases (ce qui équivaut donc à un nombre de nucléotides ou à un nombre de « lettres » dans le « code génétique »), deux unités de mesure courantes étant le kilobase (kb) et le mégabase (Mb), égaux à 1000 bases et 1 million de bases respectivement.
Un gène est une séquence d’ADN susceptible d’être transcrite en ARN. L’ARN est une séquence de nucléotides qui est la copie du code de l’ADN ”
Du coup on essaie de parler de “patrimoine génétique” , ” génome”, “génotype” quand ça s’y prête (évitant aussi l’emploi du terme “programme”). L’utilisation comme verbe ne pose pas de soucis, même s’il y a eu un petit débat un moment. Doit on dire :
– ” le gène SrY code POUR la protéine TDF ”
– ” le gène SrY code la protéine TDF ”
Il semblerait que, contrairement à l’usage, on doive plutôt dire la deuxième porposition (mais bon, ça c’est nettement moins grave qui de lire : ” dans le code génétique de l’individu, on observe une substitution bla bla bla…” qui donne autant envie de s’arracher les cheveux qu’une formulation finaliste !)
Je n’utilise bien-sûr jamais le terme “programme” : pour moi, c’est cette terminologie-là qui est vraiment trompeuse, celle du “programme génétique”, comme si le contenu de l’ADN était un programme définissant le devenir de l’organisme.
Pour moi il n’y a pas d’équivalence entre la notion de code et celle de programme. C’est “code” au sens des techniques de communication, de transmission d’information ; au sens d’un système d’équivalence entre des “signifiants” et des “signifiés”. D’ailleurs, l’usage de code/coder pour parler de programmation informatique est assez récent il me semble. De mon temps on disait programme/écrire un programme en tout cas.
Dans le passage que vous citez, ça renvoie bien à cette notion. De plus, “génotype” n’irait pas, et si je peux remplacer “code génétique” (que j’ai mis entre guillemets) par “génome”, il restera la première occurrence de “code”…
Quant à “patrimoine génétique”, je veille quant à moi au contraire à ne pas l’utiliser car c’est sexiste… 😉
Pour ce qui est de “coder pour” je sais que ça n’est pas jugé très correct mais si on revient au sens du verbe “coder”, je trouve que c’est encore plus incorrect de dire “coder la protéine X”.